До 2022 года варианты SARS-CoV-2, вызывающие новые волны COVID-19, возникали примерно с полугодовыми интервалами. И была надежда на то, что после очередной волны, вызванной вариантом Дельта, наступит какое-то затишье. Тем более что в некоторых странах к началу этой волны было вакцинировано уже почти всё население. Но неожиданно быстрое распространение варианта Дельта принесло две новости – плохую и хорошую. Плохая новость заключалась в том, что иммунизация любыми нынешними вакцинами плохо защищает от инфицирования этим новым вариантом вируса. А если и защищает, то ненадолго. Хорошая – это то, что всё-таки защищает, но преимущественно от развития тяжёлых форм пневмонии. И существенно снижает летальность. В результате массовая вакцинация и увеличение количества переболевших почти не влияют на распространение вируса, но неплохо снижают смертность от COVID-19.
Появление варианта Омикрон в целом не изменило ситуацию. Но влияние вакцинации уменьшилось, а сочетание имеющегося коллективного иммунитета с большей контагиозностью и меньшей летальностью самого вируса вселило надежду на то, что очередная волна эпидемии станет более массовой, но менее кровожадной. Т.е. сможет сформировать коллективный иммунитет со сравнительно небольшими потерями населения, что позволит надеяться на лучшее – на переход эпидемии в тлеющий режим, не требующий экстраординарных мер от здравоохранения. При этом готовиться нужно к худшему – к появлению новых высококонтагиозных вариантов SARS-CoV-2 и к «штормовому» течению пандемии, при котором последовательность новых волн эпидемии утратит сезонную упорядоченность и они начнут чередоваться всё чаще и чаще, накладываясь друг на друга.
«Штормовой» прогноз вытекает не столько из необычайно высокого количества мутаций (37) в несущем иммунопротективные эпитопы S-белке варианта Омикрон, сколько из наличия замены изолейцина на валин (I42V) в экзонуклеазном домене белка NSP14, отвечающем за корректировку ошибок коронавирусной РНК-полимерзы. Все предыдущие варианты вируса, получившие широкое распространение, содержали не больше дюжины аминокислотных замен в S-белке и обладали нативной корректирующей экзонуклеазой, обеспечивающей высокую консервативность вирусного генома.
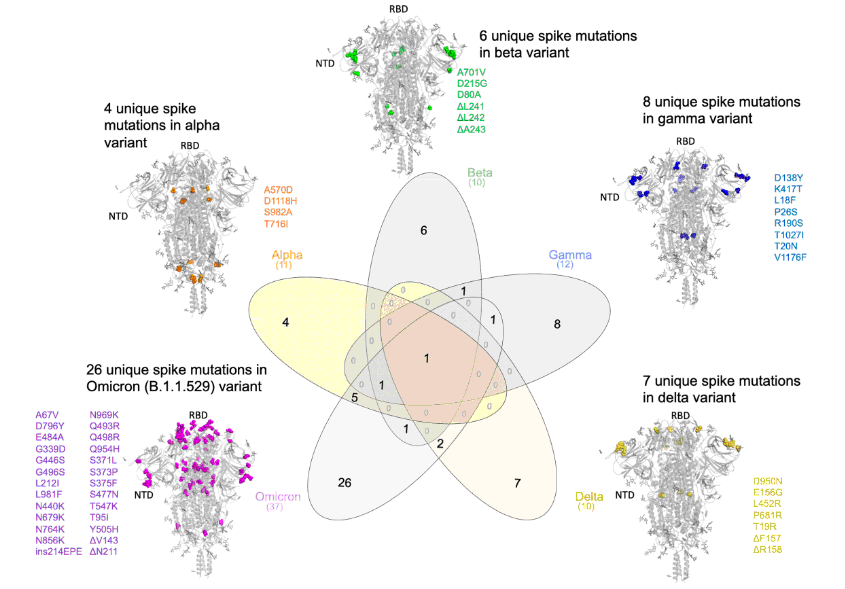
На диаграмме Венна видно, что пятёрка наиболее распространённых вариантов SARS-CoV-19 имеет в гене S только одну общую мутацию, отличающую их от референсного (Уханьского) варианта. И что Омикрон наиболее близок варианту Альфа (6 общих мутаций). Имеются перекрывания и с другими вариантами, но всего по одной (Бета и Гамма) или по двум (Дельта) одинаковым мутациям. При наличии 26 уникальных мутаций такие перекрывания можно считать незначительными.
Причиной появления мутаций в гене S, как правило, является селективное давление иммунной системы, приводящее к накоплению аминокислотных замен в иммунопротективных эпитопах спайк-белка. Поэтому для определения локализации таких эпитопов можно использовать анализ генетической вариабельности участков гена S. Исходные данные для такого анализа можно взять из таблицы, представленной на сайте сингапурского Агентства науки, технологий и исследований (A*STAR).
В этой таблице (специализированной базе данных) приведены частоты всех мутаций спайк-белка, которые можно обнаружить в GISAID — самой крупной базе данных геномных последовательностей SARS-CoV-2. Если представить табличные данные в виде графика, показывающего порядковые номера аминокислот в белке (по оси X) и количество аминокислотных замен (по оси Y), то получится что-то вроде линейчатого спектра излучения, линии которого соответствуют мутациям наиболее распространённых вариантов вируса. Частоты таких мутаций могут измеряться сотнями тысяч, а иногда и миллионами. Но если все самые крупные значения частот (>10 000) приравнять к 10 000, то линейчатость сохранится, но график станет более информативным. Особенно если позиции мутаций сопоставить с доменной структурой S-белка.
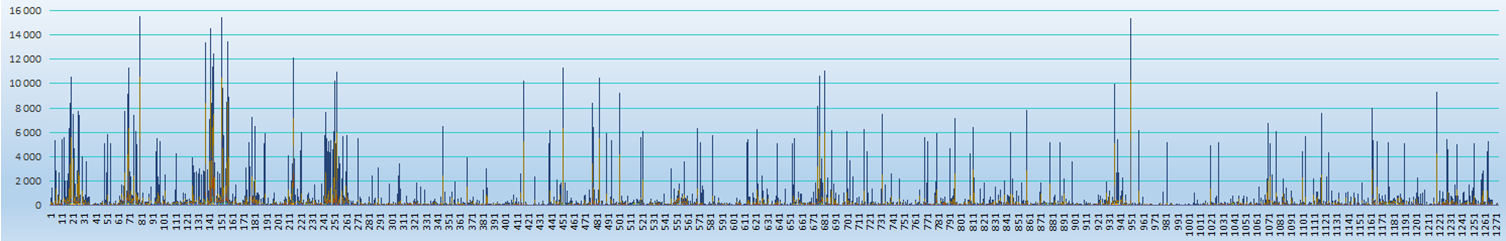
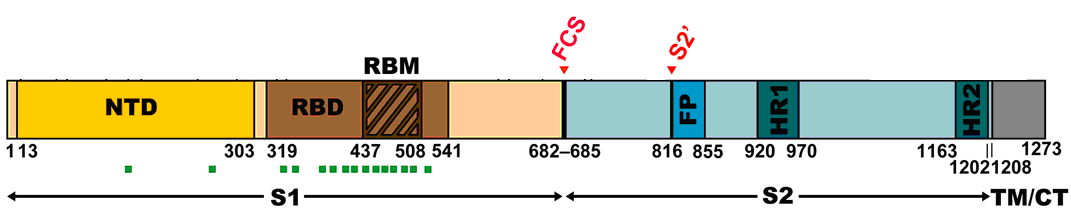
Обычно считается, что нейтрализующие вирус антитела должны взаимодействовать с рецептор-связывающим доменом RBD (Receptor Binding Domen – 319-541). Поэтому многие иммуноферментные тест-системы определяют наличие в крови иммуноглобулинов, узнающих только RBD. Но на графике видно, что наиболее вариабельные (иммунопротективные) эпитопы S-белка сконцентрированы в его N-терминальном домене (NTD – 13-303). Поэтому проверка наличия в крови иммуноглобулинов на SARS-CoV-2 самыми «крутыми» импортными тест-системами типа Architec (Abbot, США), выявляющими антитела только на RBD, после перенесённого COVID-19 или вакцинации может давать отрицательный результат.
Сравнительно невысокая вариабельность RBD может объясняться повышенной консервативностью аминокислот, отвечающих за взаимодействие с клеточным рецептором. Причём самым консервативным должен быть RBM (Receptor Binding Motiv – 437-508), но и в нём возможны аминокислотные замены, препятствующие связыванию некоторых моноклональных антител, а также ослабляющие или усиливающие связывание вируса с рецептором ACE2.
На карте гена S, представленной на сайте Национального центра
биотехнологической информации (NCBI), рецептор-связывающий мотив (RBМ) включает 71 аминокислоту.

Из них только 20 там отмечены как непосредственно взаимодействующие с клеточным рецептором ACE2 (выделены жирным шрифтом). Они должны отличаться повышенной консервативность, но похоже на то, что эти данные требуют уточнения. Во всяком случае, для варианта Омикрон. У него в RBM сконцентрирован целый десяток аминокислотных замен (отмечены сиреневым шрифтом), причём одна из них (Q493R) расположена в гиперконсервативном гептапептиде YFPL(Q/R)SY. Уже известно, что эта замена ухудшает связывание вируса с клеточным рецептором, но неподалёку имеется мутация (N501Y), способная усиливать такое связывание. Последствия такого сочетания мутаций пока непредсказуемы.
Таблицу с полным перечнем и сопоставлением мутаций двух линий варианта Омикрон (BA.1 и BA.2) можно найти здесь: https://github.com/cov-lineages/pango-designation/issues/361
В более наглядном виде расположение мутаций у этих (и у многих других) вариантов SARS-CoV-2 представлено на сайте Стэнфордского университета.
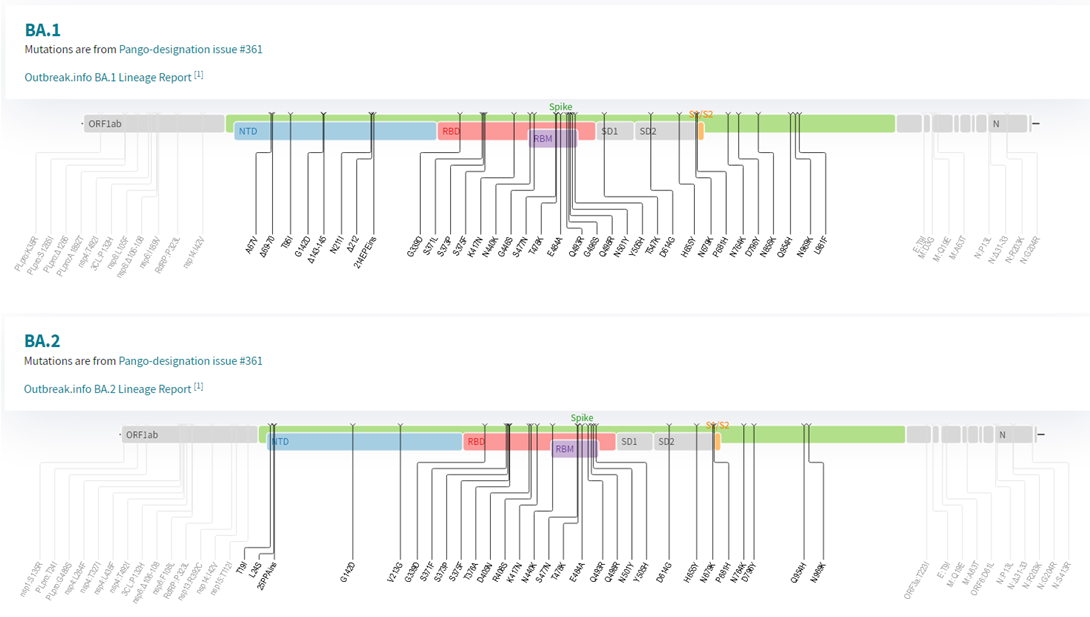
Линия BA.2 имеет много отличий от BA.1, но их объединяет обилие мутаций и присутствие аминокислотной замены I42V (замена нуклеотида A18163G ) в экзонуклеазном домене гена nsp14, отвечающем за коррекцию ошибок РНК-полимеразы.
В «доомикроновские» времена мутаций у вариантов VOC (variants of concern) было недостаточно для определения иммунопротективных эпитопов (участков S-гена с повышенной вариабельностью) SARS-CoV-2 (см. предыдущую публикацию). И это вселяло оптимизм. С появлением серии омикроновских вариантов (B.1.1.529, BA.1, BA.1.1, BA.2, BA.3) надежды на возможное окончание пандемии растаяли. Зато графики вариабельности S-гена у наиболее опасных вариантов вируса стали более наглядными.
Ниже показаны результаты сопоставления выровненных последовательностей гена S вариантов A (референсный, уханьский ), Альфа (B.1.1.7), Бета (B.1.351), Гамма (P.1), Дельта (B.1.617.2) и Омикрон (BA.1 и BA.2), полученные при помощи программы Nuclon 2.0.
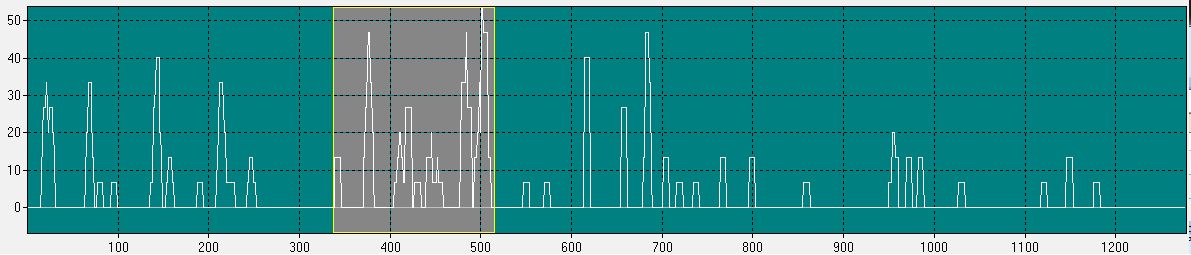
Выделенный на графике участок (серый, с жёлтой рамкой) примерно соответствует RBD (аминокислоты 337-516, смежные значения количества мутаций суммированы с рамкой 7). В текстовом окне программы Nuclon можно подробно рассмотреть все аминокислотные замены в выделенном участке (RBD) у наиболее распространённых вариантов вируса, образующих группу VOC (см. предыдущую публикацию — «Немного о вариабельности SARS-CoV-2»).
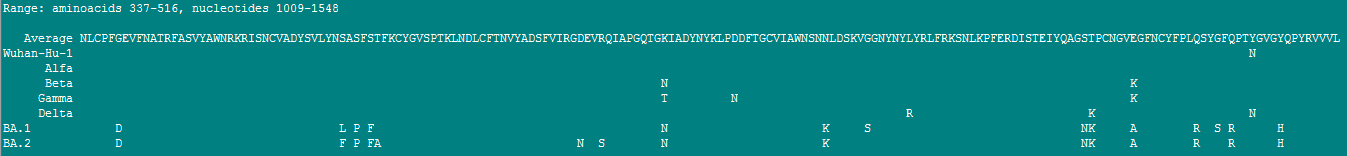
Невооружённым взглядом видно, что по количеству аминокислотных замен омикроновские варианты BA.1 и BA.2 разительно отличаются от всех прочих вариантов этой группы. Отсюда следует, что любые вакцины «уханьского» типа не могут обеспечить надёжную защиту от омикроновских вариантов SARS-CoV-2. Причём из-за быстрого чередования вариантов существующие правила проведения испытаний и регистрации новых вакцин не позволяют достаточно оперативно создавать их более эффективные разновидности. Зато анализ вариабельности гена S позволяет быстро разрабатывать тест-системы для определения новых вариантов SARS-CoV-2 методом Real-time PCR.
В конце 2021 года в прессе появились заявления представителей нескольких российских разработчиков, пытающихся зарегистрировать наборы реагентов для определения варианта Омикрон. Опередить всех конкурентов удалось Центру стратегического планирования ФМБА России, зарегистрировавшему 11 января медицинское изделие (тест-систему) под названием «Набор реагентов для выявления РНК коронавируса SARS-CoV-2 генетических вариантов Omicron и Delta на основе определения характерных для них мутаций в S гене методом полимеразной цепной реакции» («АмплиТест SARS-CoV-2 VOC v.3»).
Нуклеотидные последовательности гибридизационных зондов, используемые в подобных наборах, должны соответствовать наиболее характерным для омикроновского
варианта участкам. В публикации итальянских разработчиков (01.12.2021) в качестве зондовой последовательности были выбраны 28 нуклеотидов, перекрывающих пять мутаций гена S в гипервариабельном участке RBM (отмечены красным шрифтом, соответствуют аминокислотным заменам Q493R, G496S, Q498R, N501Y и Y505H).
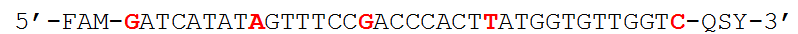
Теоретически это очень неплохой выбор. Но на практике такой зонд работать не будет из-за контактного тушения флуоресценции FAM 5’-концевым гуанином. Проще всего решить эту проблему заменой 5′-концевого FAM на ROX (с заменой 3′-концевого QSY на BHQ2), или на какой-нибудь другой флуорофор, не подверженный контактному тушению.
Ещё лучше — подобрать зонды, нацеленные не на сочетания одиночных мутаций, а на характерные для омикроновских вариантов делеции или вставки. Например, на делецию Δ69-70 или инсерцию ins214EPE в гене S. Правда, обе эти мутации отсутствуют у варианта BA.2, а делеция Δ69-70 имеется и у варианта Альфа. Но Альфа в России уже давно не встречается, а вариант BA.2 (он же – BA.1.1.529.2) в России пока не обнаружен. Судя по данным, получаемым при помощи секвенирования геномов вируса, к 16.01.2022 соотношение BA.2/BA.1 равнялось 5 203/342 240. Т.е. количество выявляемых в мире проб с вариантом BA.2 составляет ~1,5% от BA.1.
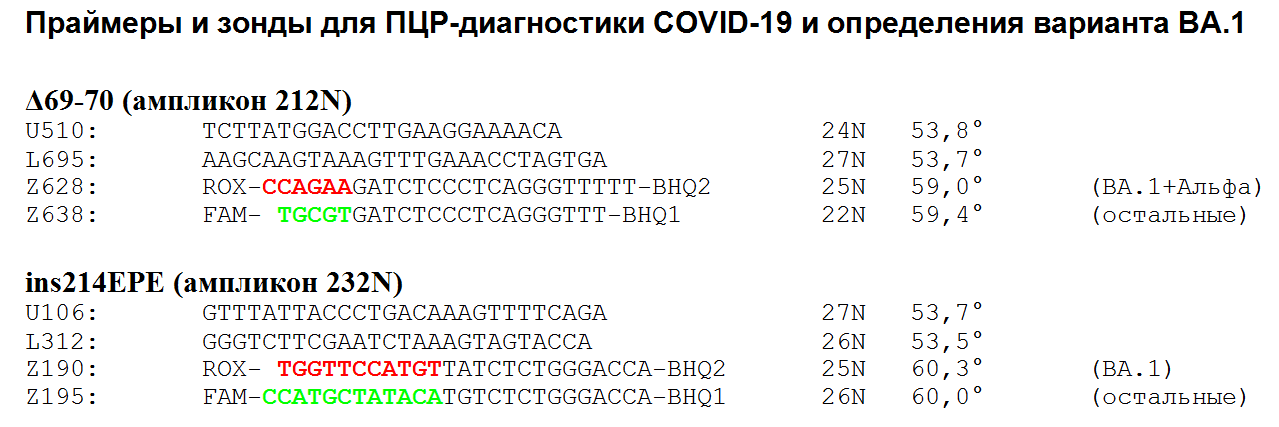
Расчёт подобных праймерных и зондовых последовательностей необходим для быстрого создания тест-систем, предназначенных для мониторинга новых вариантов SARS-CoV-2 на самой ранней стадии их распространения. Но смена превалирующих вариантов (Дельта→Омикрон) идёт настолько быстро, что к концу января производство таких тест-систем окажется неактуальным. Тем более что все (или почти все) существующие наборы реагентов для ПЦР-диагностики позволяют определять все (или почти все) варианты этого вируса. А общепринятым способом мониторинга любых новых вариантов вируса является широкомасштабное секвенирование их геномов.
Масштабы секвенирования геномов SARS-CoV-2 можно оценить по информации, регулярно обновляемой на сайте Китайского национального центра биоинформатики (CNCB).
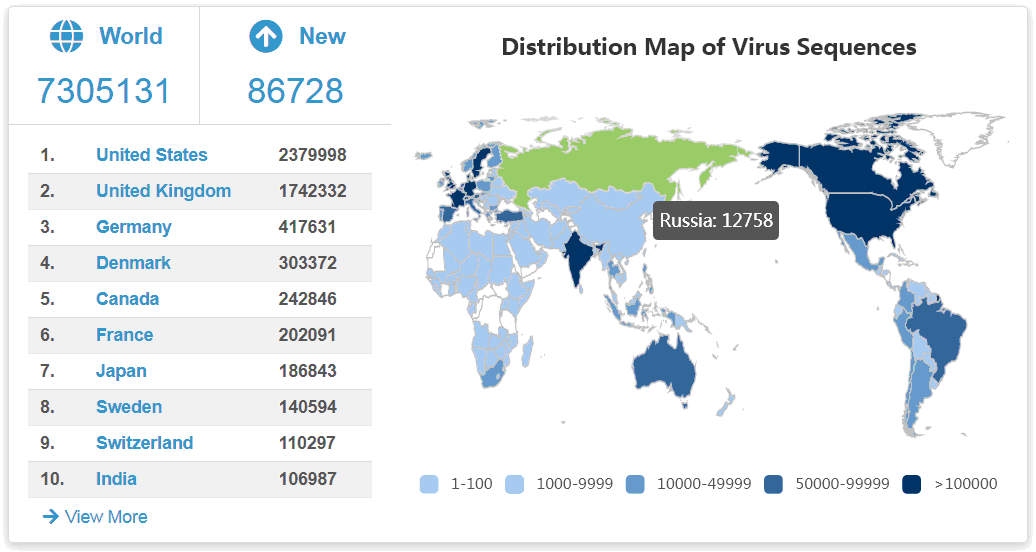
Судя по представленным там данным, к середине января открытые базы данных содержали больше 7 миллионов записей коронавирусных геномов. Тройка лидеров включает США (2 379 998 геномов), Великобританию (1 742 332 генома) и Германию (417 631 геном), а в десятку лидеров входят страны, представившие в международные базы данных больше 100 тысяч геномных последовательностей.
Перемещение курсора по интерактивной карте позволяет посмотреть количество геномов, секвенированных в разных странах. Если курсор навести на Россию, то этот показатель будет равен 12 758 (на 17.01.2022). Цифра явно занижена, поскольку не включает информацию из курируемой Роспотребнадзором базы данных VGARus, в которую к 09.01.2022 было загружено 36 715 полногеномных последовательностей и 26 786 последовательностей гена S.
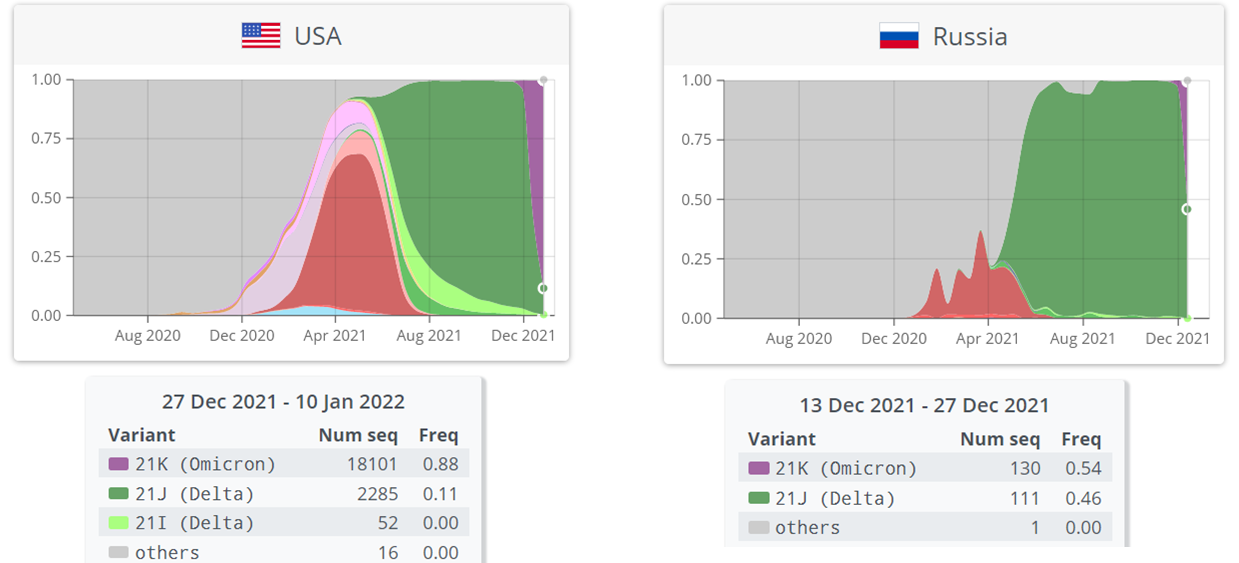
Мониторинг геномных данных позволяет следить за динамикой распространения различных вариантов вируса как в мире, так и в отдельных странах. Наиболее наглядно такая динамика представлена на интерактивном сайте CoVariants (https://covariants.org/). Правда, для построения графиков там используется только база данных GISAID (https://www.gisaid.org/). И не используются данные из VGARus (https://genome.crie.ru/).
Непредсказуемость эволюции SARS-CoV-2 стимулировала развитие массового секвенирования геномов этого вируса. А заодно улучшила финансовые показатели лидеров рынка NGS (next generation sequencing). Например, в четвёртом квартале 2021 года выручка компании Illumina благодаря рекордным продажам расходных материалов и секвенатора NovaSeq достигла 1,19 миллиардов долларов (за год – $4,52 млрд.). А в этом году в продажу поступит NovaSeq Dx – клиническая версия самой высокопроизводительной у Illumina платформы NGS-секвенирования (https://pcr.news/stati/jp-morgan-healthcare-2022-den-1-illumina/).
Для России основной проблемой, ограничивающей приобретение американских приборов и расходных реагентов, является их высокая стоимость. Дешевле (на 10%) покупать аналогичные секвенаторы (DNBSEQ-T7, DNBSEQ-G400, DNBSEQ-G50) в Китае. Ещё дешевле может стоить первый российский NGS-секвенатор (Нанофор СПС), производство которого должно начаться в ближайшие месяцы.

В 2022 году лидером рынка NGS может стать британская компания Oxford Nanopore Technologies (ONT), которой недавно удалось повысить точность нанопового секвенирования до 98% (при однократном чтении). И сделать дешёвые (от $1000) нанопоровые секвенаторы конкурентоспособными во всех областях.
Первая геномная последовательность варианта Омикрон, появившаяся в GenBank 30.11.2021 (OL672836), была получена при помощи MinION – самого дешёвого нанопорового секвенатора, отличающегося от громоздких оптических приборов исключительной лёгкостью (78 грамм) и устойчивостью к вибрациям. Последнее качество позволило компании ONT в начале 2021 года оборудовать несколько мобильных лабораторий, занимающихся геномным мониторингом вариантов SARS-CoV-2 на просторах Великобритании.

В России о подобном мониторинге можно только мечтать (см. «Задачи отечественного секвенаторостроения»). И стараться не переживать из-за появления новых вариантов SARS-CoV-2 и вызываемых ими волн COVID-19. Тем более что таких волн будет всё больше и больше.
«В шкафу нашел маску и грустно улыбнулся, поправляя скафандр» © О.Огарков


