Кто не любил в детстве собирать конструкторы? Я до сих пор помню эту красную коробочку с кучей металлических деталей, инструментов и морем возможных результатов лишь бы была фантазия, время и желание. LEGO тоже не стоит забывать, хоть тут все было чуть проще и гораздо красочнее. Но вот что сложнее и того и другого так это конструкторы наноструктур на базе ДНК-оригами. До сих пор все «детальки» подобных конструкций моделировались вручную, что занимало уйму времени и сил. Представьте, что все детали LEGO вам нужно создавать самому, прежде чем собрать их в гигантского робота с лазерами, реактивными двигателями и пулеметом на плече. Но что-то детские воспоминания увели нас не в ту степь.
Шутки в сторону. Сегодня мы с вами познакомимся с алгоритмом, позволяющим автоматически создавать ДНК-оригами достаточно разнообразной формы. Ранее изменение формы нитей ДНК до необходимой выполнялось вручную, что сильно ограничивало возможности подобной процедуры. Данный же алгоритм позволяет создавать детали ДНК конструктора автоматически, что позволяет в дальнейшем использовать их для формирования двумерных и трехмерных наноструктур. Плюс этот алгоритм доступен всем желающим. Доклад исследовательской группы поможет нам разобраться что и как работает. Поехали.
Основа исследования
Использование ДНК как строительного материала в наноструктурах широкого спектра применения за последние годы стало целью множества исследований и экспериментов. Многие из них добились отличных результатов, но как мы знаем — нет предела совершенству. В качестве примера исследователи приводят монодисперсные ДНК структуры в мегадальтонном масштабе с высоким коэффициентом свертывания, что было реализовано посредством длинных каркасов из ssDNA (single-stranded DNA) — одноцепочечных/однонитевых ДНК. Однако в данном случае именно длина нитей ДНК является основным ограничением размера единой ДНК-оригами. Это ограничение можно обойти формированием структуры, состоящей из нескольких оригами сразу.
Также мы с вами уже знаем (Крестики-нолики: демонстрация управляемого процесса реконфигурации ДНК структур), что некоторые ДНК структуры возможно создавать посредством ДНК плиток, что позволяет формировать сложную геометрию этой самой структуры.
Но опять же все упирается в то, что процесс выполняется вручную, а это долго и муторно. А что любят ученые? Правильно, автоматизацию ну или хотя бы частичную автоматизацию. Подобные инструменты уже имеются (пример ученых — caDNAno или Tiamat), однако эти программы имеют свои недостатки. Во-первых, все же ручное создание каркаса. Во вторых, ограниченность вышеупомянутых алгоритмов в вопросе систематизации проектирования геометрии структуры.
Если в двух словах, то в данной работе исследователи поставили перед собой цель создать алгоритм, который будет практически все делать самостоятельно. Таким образом любой человек может задать только параметры внешней оболочки желаемой структуры, а программа заполнит внутреннюю часть самостоятельно всеми необходимыми ДНК нитями, скобами, последовательностями и прочим. Также можно процесс провести и в обратном порядке — задать все «внутренности», так сказать сетку структуры, а алгоритм построит правильный и самый оптимальный каркас вокруг нее.
Данная программа (PERDIX) с открытым исходным кодом доступна абсолютно всем, за что спасибо исследователям. Таким образом, любой желающий может не только «поиграться» с самой программой, но и, возможно, внести некие совершенствующие правки в ее код. Где ее скачать и как запустить мы рассмотрим в конце статьи.
А теперь давайте рассмотрим примеры применения данной программы, которые нам предоставила исследовательская группа.
Результаты проверки PERDIX
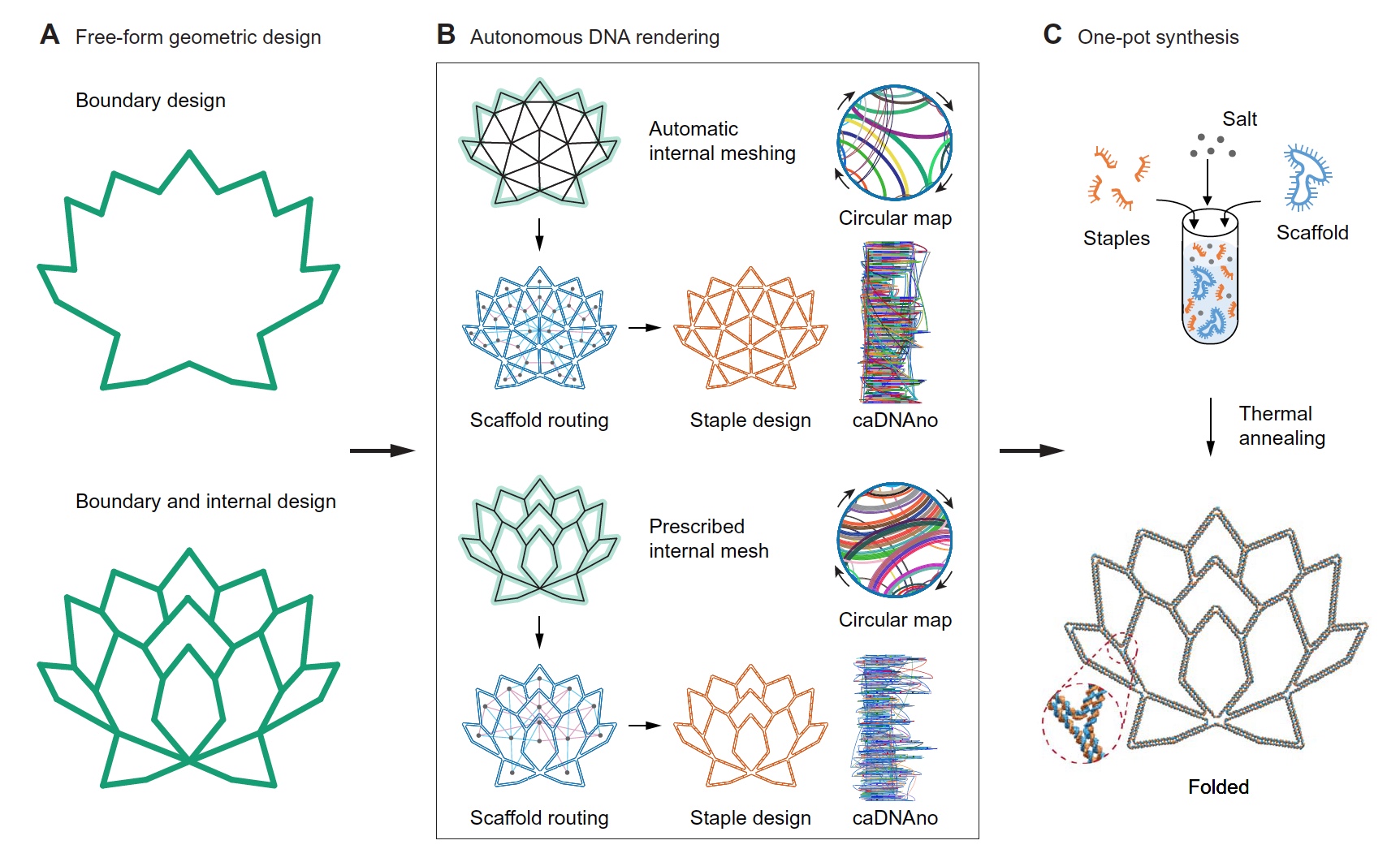
Изображение №1
В качестве вводных данных используется либо изображение границы желаемой структуры (1А, сверху), либо рисунок с более точными геометрическими деталями (1А, снизу). Далее уже сама программа заполняет внутреннее пространство.
Когда задается только «очертание», внутренняя геометрия сетки структуры формируется за счет треугольников посредством программы с открытым кодом DistMesh, которая требует введения только плотности сетки, как необходимого параметра.
Когда же задана форма и геометрические линии (1А, снизу), используется Shapely — пакет Python для манипулирования и анализа плоских геометрических объектов. Shapely генерирует полигональную сетку, в которой ее линии и точки их пересечения формируют необходимую геометрию каркаса.
Далее, после определения нужной формы, необходимо было задать номинальную длину ребра, которая должна быть ≥ 38 bp (спаренных оснований) или же 12.58 нм. Таким образом в необходимой структуре однозначно будут присутствовать минимум два парных кроссинговера на каждое ребро.
Затем все целевые линии будущей структуры преобразуются в DX ребра, у которых все вершины являются многоветочными точками соединения (1В). Следуя принципам теории графов, автоматически формируется оптимальная структура каркаса, после чего завершается процесс назначения последовательности дополнительных ДНК-скоб. Процесс ручного редактирования выходных данных (результатов) осуществляется посредством сгенерированного caDNAno файла.
Очень важной особенностью своей системы ученые называют факт того, что ребра не обязательно должны равняться целому числу двойных спиралей В-ДНК (10.5 bp). А это позволяет получить больше свободы в проектировании геометрии структуры.
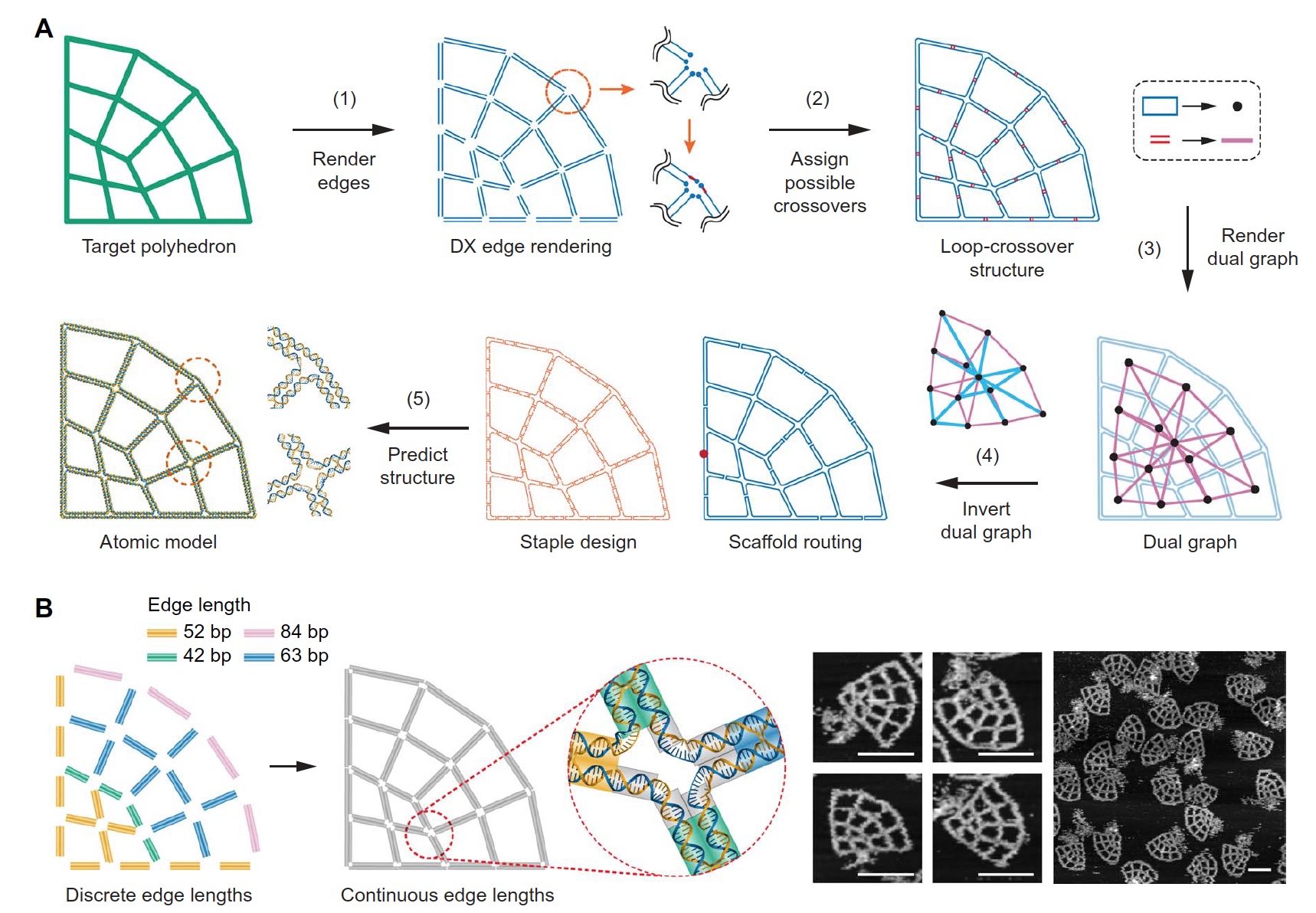
Изображение №2
Дабы быстро и точно выполнить автоматическую часть процесса формирования геометрии структуры все ребра сначала необходимо было преобразовать в DX motifs посредством рендеринга каждого ребра целевой геометрии с использованием двух антипараллельных линий каркаса (2А). Далее они объединяются у единой вершины, что делает каждое ребро частью петли. А это в свою очередь приводит к формированию единой большой внешней петли, состоящей из малых петель. Следующим шагом является нахождение всех возможных вариантов кроссинговеров между соседствующими петлями, то есть формирование структуры пересечения петель.
Третьим шагом в этом процессе является представление всех полученных структур как совокупности узлов, а всех кроссинговеров как ребер будущей структуры.
Далее вычисляется покрывающее дерево двойного графа, после чего оно конвертируется в ssDNA каркас. Этот процесс происходит полностью автоматически. И наконец, происходит генерация трехмерной структурной модели на атомном уровне.
На изображении 2В продемонстрировано два варианта формирования необходимой геометрии структуры: с дискретной длиной ребра или с непрерывной длиной ребра. Второй вариант позволяет создавать структуру с произвольной длиной ребра, что в свою очередь дает возможность получить наибольшее свертывание. Данные вариации структурирования геометрии были проверены посредством сканирующего атомно-силового микроскопа (снимки справа на изображение 2В).

Изображение №3
Исследователи также поставили перед собой цель более детально рассмотреть такие аспекты целевой геометрии как механическая жесткость, точность формы, размер структуры, размер внутренней сетки и типы ее элементов. Также необходимо было понять насколько важную роль играют N-ветви, то есть число соединений между ребрами и, как результат, общая сложность структуры. Анализ данных АСМ показали, что двумерные структуры с большим числом ветвей и большей длиной ребер (за счет неспаренных нуклеотидов) получаются достаточно хорошо сформированными, а неоднородность частиц в их структуре минимальна.
Внутренняя сетка структуры имела три варианта: треугольники с одинаковым направлением, квадраты и треугольники с разными направлениями (3С). Именно модель с треугольниками показала наибольшую точность в формировании необходимой геометрии, в отличие от модели с квадратами. Наличие смешанных направлений в треугольной модели, так сказать, позволяло получить более симметричные N-ветви и более точную форму (менее искаженную).
Ученые отмечают, что анализ внутренней сетки моделируемой структуры крайне важен для понимания механической жесткости этой структуры. И именно описанная выше модель идеального подходит для создания достаточно прочных, точных (в плане соответствия заданным параметрам) и гибких структур.

Изображение №4
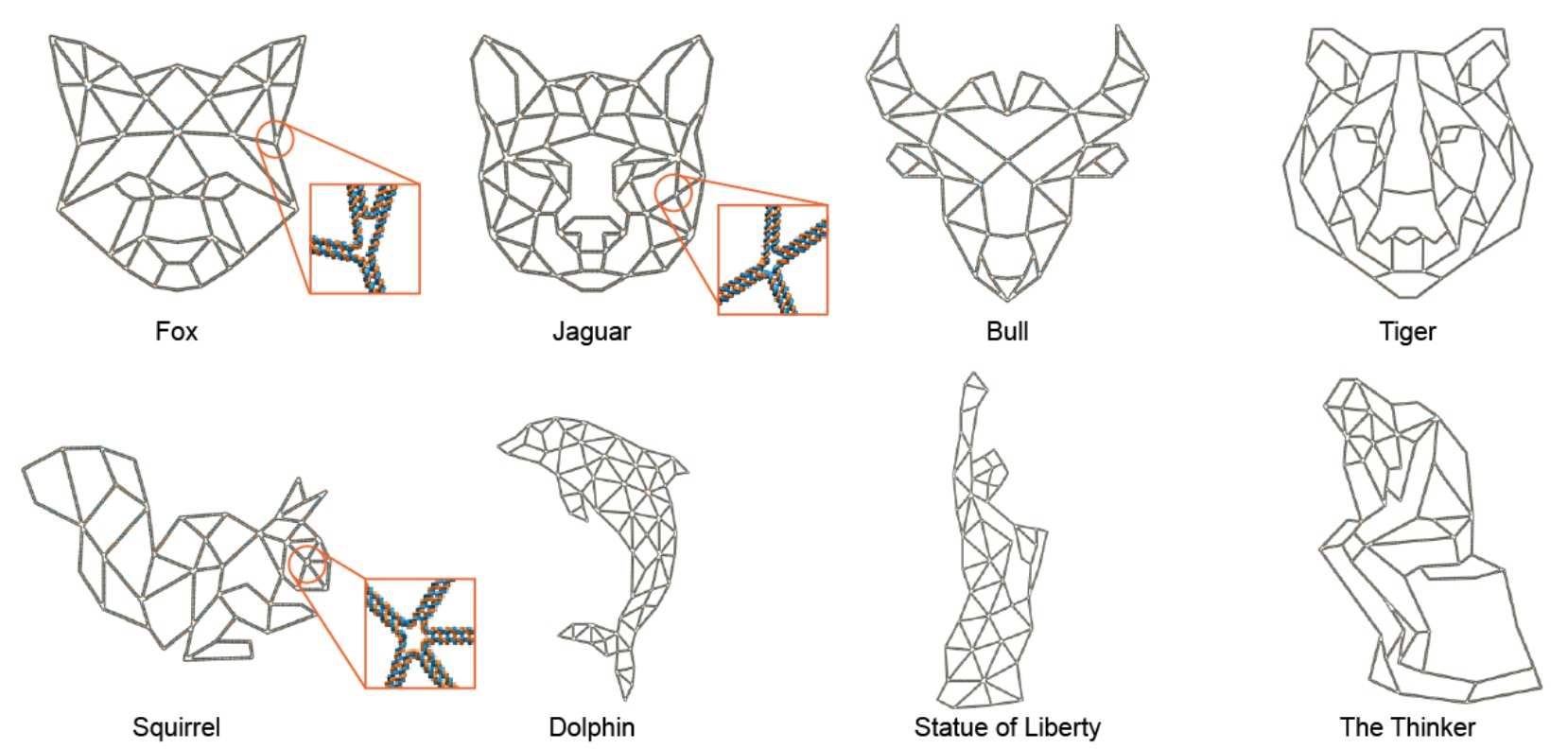
Конечно же ученые не лишены чувства прекрасного, так сказать. Посему ими было решено продемонстрировать возможности своей разработки путем проектирования структур 15 разных форм, точнее с разными внутренними сетками и каркасом. Варианты мы можем видеть на изображении выше: тут и «обычные» квадраты, и четверть круга, и даже лотос.
Программное обеспечение с открытым исходным кодом
Как я уже упоминал ранее, труд исследователей открыт всем желающим по ссылке (), где вы можете найти необходимые программные элементы (MATLAB, Python 2.7 и Shapely 1.6.4).
Что касается самого алгоритма формирования геометрии структур ДНК-оригами, то инструкция по его скачиванию и запуску представлена ниже в формате видео. Все ролики я спрячу под спойлер, чтобы не растягивать статью.
Для более детального ознакомления с данным исследованием и его результатами настоятельно рекомендую заглянуть в доклад исследовательской группы и дополнительные материалы к нему.
Эпилог
Данное исследование было нацелено на упрощение достаточно затратного по времени и силам процесса. И это получилось. Алгоритм PERDIX позволяет создавать структуры с самой разной геометрией, при этом задавая минимум необходимых параметров. ДНК уже давно стали объектом изучения ученых не только как часть всего живого и носитель информации, но и как возможный вариант основы технологий будущего. Подобные труды позволяют более детально понять (причем наглядно) насколько широки возможности наноструктур, созданных на базе ДНК-оригами.
Второй приятный момент в этом исследовании, по крайней мере для меня, это то, что алгоритм доступен всем желающим. Любой может им воспользоваться, любой может его усовершенствовать. Давая полный доступ как к своему докладу, так и к самому программному обеспечению, ученые не только способствуют популяризации своей научной отрасли, но и науки как таковой.
Я встречал статьи, в которых научные деятели достаточно критично высказывались насчет платной системы доступа к докладам, утверждая что это ставит дополнительную преграду между знаниями и простыми обывателями. Ситуация в этом вопросе очень неоднозначная и спорная, ибо зарабатывать должны все (и научные издания, и исследовательские группы, и научные сайты), но и сильное удорожание доступа к некоторым исследованиям делают их недоступными для простых любителей науки, студентов и даже для некоторых профессоров и научных деятелей. Как я уже сказал, вопрос неоднозначный, посему сильно затрагивать его не будем. В любом случае, радует, что сегодняшние наши герои предоставили нам полный доступ к своему труду, который заслуживает не только внимания, но и откровенного восхищения.
Благодарю за внимание, оставайтесь любопытствующими и хорошей всем рабочей недели, ребята.
Спасибо, что остаётесь с нами. Вам нравятся наши статьи? Хотите видеть больше интересных материалов? Поддержите нас оформив заказ или порекомендовав знакомым, 30% скидка для пользователей Хабра на уникальный аналог entry-level серверов, который был придуман нами для Вас: Вся правда о VPS (KVM) E5-2650 v4 (6 Cores) 10GB DDR4 240GB SSD 1Gbps от $20 или как правильно делить сервер? (доступны варианты с RAID1 и RAID10, до 24 ядер и до 40GB DDR4).
VPS (KVM) E5-2650 v4 (6 Cores) 10GB DDR4 240GB SSD 1Gbps до весны бесплатно при оплате на срок от полугода, заказать можно тут.
Dell R730xd в 2 раза дешевле? Только у нас 2 х Intel Dodeca-Core Xeon E5-2650v4 128GB DDR4 6x480GB SSD 1Gbps 100 ТВ от $249 в Нидерландах и США! Читайте о том Как построить инфраструктуру корп. класса c применением серверов Dell R730xd Е5-2650 v4 стоимостью 9000 евро за копейки?
Источник

