
С учетом того, что закон Мура является всего лишь эмпирическим наблюдением и упирается в физическую вместимость микропроцессора, то есть, в количество транзисторов, которые можно уместить на единицу площади, вполне логично, что программно-аппаратная инженерия пытается уйти от традиционных носителей информации на материале соединений кремния. Тем более, что срок действия закона Мура явственно подходит к концу. Возможной альтернативой для вычислительной неорганики много лет мыслится вычислительная органика. То есть, теоретически, а также (возможно) практически должны быть варианты хранения информации в белках и нуклеиновых кислотах. Тем более, что нуклеиновые кислоты в природе превосходно справляются с кодированием и передачей информации.
Сразу оговоримся, что для информации нужно не только хранилище; нужен еще и процессор, а также устройства ввода-вывода. Поскольку до создания подобной инфраструктуры еще очень далеко, тема казалась бы спекулятивной, но в январе 2021 года в журнале «Nature of Chemical Biology» была опубликована статья, описывающая довольно простую технологию кодирования 3-битных информационных последовательностей в ДНК. Вот о чем она.
Двойная спираль. Винтовая лестница в обход закона Мура
В современном мире постоянно генерируется все больше данных, и исследователи как могут изобретают новые способы их хранения. ДНК по-прежнему считается весьма перспективной в качестве исключительно компактного и устойчивого носителя информации. А прямо сейчас формируется новый подход, позволяющий записывать цифровые данные непосредственно в геномы живых клеток.
Попытки переориентировать технологии запоминания данных, изобретенные природой, не новы, но в последнее десятилетие интерес к таким подходам оживился, и уже есть заметные достижения в этой области. Ситуация вызвана взрывным ростом генерируемых данных, причем, нет никаких признаков его замедления. Предполагается, что в 2025 году во всем мире ежедневно будет создаваться 463 эксабайт данных.
Хранение всех этих данных с применением кремниевых технологий вскоре может стать непрактичным, но выход может заключаться в использовании ДНК. Во-первых, плотность информации ДНК в миллионы раз выше, чем на обычных жестких дисках. Всего в одном грамме ДНК можно хранить до 215 миллионов гигабайт данных.
Кроме того, при правильном хранении ДНК исключительно стабильна. В 2017 году ученым удалось полностью восстановить геном лошади (вымершего вида), жившей 700 000 лет назад. Научившись хранить данные и обращаться с ними на том же языке, который используется в природе, мы открываем путь к множеству новых биотехнологических возможностей.
Основная сложность заключается в том, чтобы найти интерфейс между цифровым миром информатики и биохимическим миром генетики. В настоящее время для этого требуется синтезировать ДНК в лаборатории, и этот процесс по-прежнему дорогой и сложный, хотя, стоимость синтеза ДНК быстро снижается. Полученные последовательности затем тщательно хранятся in vitro, пока не потребуется вновь к ним обратиться, либо их можно внедрять в живые клетки при помощи технологии CRISPR, предназначенной для редактирования генов.
CRISPR – это популярная новая технология редактирования геномов, именно за нее была вручена Нобелевская премия по химии в 2020 году. Аббревиатура CRISPR означает «короткие палиндромные повторы, регулярно расположенные группами». Подробнее о ней можно почитать в замечательной свежей статье на Хабре. Здесь же оговоримся, что в интересующем нас контексте криспры могут использоваться так: бактерий стимулируют электрическим сигналом, заставляя таким образом вставлять в ДНК заранее определенные последовательности, соответствующие нулям и единицам. Статья об этом была опубликована 24 января 2021 года. Отметим, что применение электрических сигналов для встраивания криспров – это инновационный метод, ранее применялись только биохимические взаимодействия, например, индуцируемые фруктозой. Кроме того, хранение информации в клетках кишечной палочки исключительно эффективно – согласно более раннему источнику, в клетках кишечной палочки можно зашифровать 1019 бит информации на кубический сантиметр. При дальнейшей экстраполяции можно вычислить, что ДНК, необходимую для хранения всех данных, имевшихся в распоряжении человека на 2017 год, можно уложить в виде куба с гранью 1 м.
Остановимся подробнее на описании этого эксперимента.
Авторы заменили систему ввода, использующую индуцирование фруктозой на систему электронного ввода, которая позволяет кодировать более длинные последовательности. При помощи синтетической адаптивной системы CRISPR, работающей по принципу редокс-отклика, удалось усилить экспрессию генов в ответ на рост электрического напряжения. Таким образом цифровые данные были закодированы непосредственно в бактерии, без необходимости синтеза ДНК in vitro.
Воспользовавшись 3-битным потоком данных, исследователи за три последовательных круга операций смогли внедрить в клетку различные комбинации разрядов в виде массивов CRISPR, которые, будучи внедрены в бактериальную ДНК, снабжались уникальными ID. Такие штрих-коды позволили записать в клетки E.Coli 72-битное сообщение ‘Hello world!’
Данное сообщение с 90-процентной точностью воспроизводилось в клетках после деления на протяжении 80 поколений. Соответственно, это надежная стратегия для хранения, амплификации и восстановления данных в живых клетках, позволяющая обойтись без сложных систем хранения ДНК. Бактериальные клетки значительно более устойчивы к повреждениям, чем сырая ДНК – это удалось доказать, поместив такие измененные бактерии в почву, а затем взяв их оттуда и вновь считав закодированные данные.
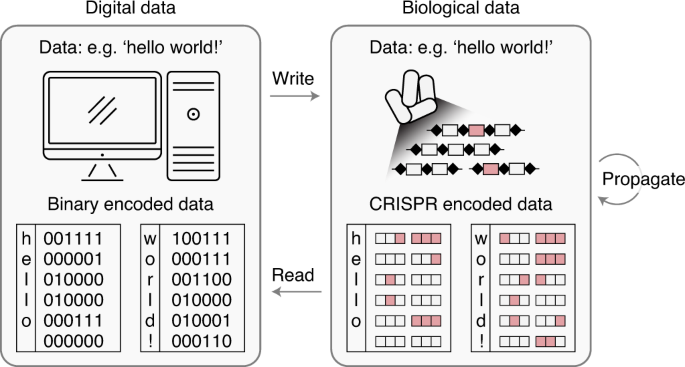
Итак, бактериальный биоматериал и молекула ДНК в частности удобны для хранения любой информации, а не только биологической. Кроме того, размножение бактерий решает проблему с резервным копированием данных, а информационная плотность лабораторной бактерии E.Coli более чем достаточна с учетом потребностей современной информатики.
Мембранные вычисления
Ниже в этой статье мы также затронем тему переноса логических операций из информатики на биоматериал, но до этого поговорим о концептуальном сходстве программирования и биологии, выраженном в концепции «мембранных вычислений». Здесь предлагается введение в мембранные вычисления (презентация). Концепция мембранных вычислений, впервые сформулированная в 1998 году, предполагает, что выстраивание программных систем и алгоритмов возможно по такому же восходящему принципу, что и формирование организма из отдельных клеток и тканей. Мембранные вычисления предполагают операции над группами (мультимножествами) объектов, каждое из которых логически находится в пределах мембраны, напоминающей клеточную, и может обмениваться данными с другими подобными мультимножествами. Мультимножества могут группироваться в совокупности, напоминающие биологические ткани, органы, либо колонии бактерий. При этом подобные структуры удобны для представления «клеток» (мультимножеств) в качестве узлов графа, а также хорошо поддаются распределенным и параллельным вычислениям. Возможные области применения парадигмы – биология, биомедицина, лингвистика, компьютерная графика, экономика, оптимизация, криптография.
Наряду с «клеточными» и «обычными тканевыми» мембранными системами наиболее интересны импульсные нейронные P-системы (spiking neural P-systems), которые сближаются по структуре с нервной тканью. В вышеприведенной презентации они рассматриваются на слайде 93 и далее и к 2020 году уже находят реальное практическое применение: например, в распознавании образов. Учитывая, что мембранные системы располагают к разработке специализированных эволюционных алгоритмов, в частности, для целей оптимизации, можно говорить о целом классе наработок, которые естественным образом подходят для реализации на молекулярно-биологическом материале.
Масштабирование вычислений и другие прикладные проблемы
Кроме того, принципиальное значение для традиционной вычислительной техники играют:
1. Оперативная память
2. Масштабирование вычислений
3. Булева логика
В статье 2017 года, подготовленной в MIT (Массачусетском технологическом институте) рассказано, как эти концепции могут быть реализованы на уровне биохимии. Память является фундаментальным элементом вычислительных систем, так как позволяет учитывать не только историю произошедших событий, но и события, происходящие в системе и ее окружении прямо сейчас. Масштабирование позволяет справляться с распределением вычислений, причем, в рассматриваемой области достигается простым увеличением числа клеток. Логику операций в ДНК можно закладывать на уровне сайтов присоединения, на месте которых ферменты-рекомбиназы могут вставлять, вырезать или переворачивать фрагменты ДНК, в зависимости от запрограммированной операции. Также при помощи ферментов можно программировать в клетке поведение, аналогичное работе машины состояний. Еще в 2013 году было показано, что при помощи ферментов в ДНК можно реализовать все логические операции, не создавая при этом каскадов логических вентилей. При этом отдельно придется программировать порядок действия ферментов, которые будут воздействовать на ДНК. Существуют исследования, описывающие разработку биологических машин состояний на основе рекомбинации ДНК. Для ввода и вывода в таких системах используются искусственно сформированные плазмиды, при внедрении которых в клетку эта клетка должна перейти в заданное состояние. Точность воспроизведения заданного состояния даже через три поколения близка к 100%.
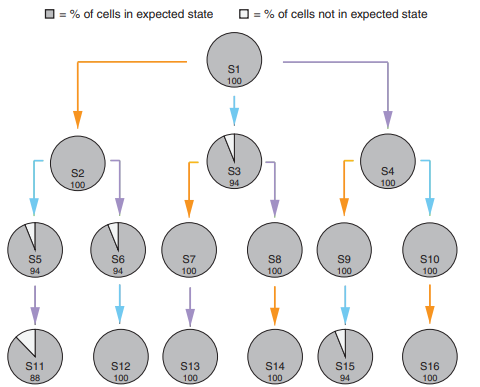
Что касается возможных проблем такого подхода в продакшене – зависимость скорости операций, например, считывания данных, от скорости транскрипции белка. Также процесс передачи информации в живой клетке невозможно полностью защитить от мутаций, а скорость накопления мутаций, по-видимому, должна повышаться с ускорением работы таких эволюционных механизмов.
Интересно, что работа по проектированию логических вентилей на уровне белков проводится и для решения чисто биологических задач, например, для борьбы с истощением запаса иммунных Т-клеток. Авторы этой работы подчеркивают, что белки в качестве логических единиц могут работать и без клеточного аппарата, непосредственно в межклеточной среде, а синтез белков с заданными свойствами позволяет управлять и топологией их молекул, и принципами связывания с другими молекулами. Кроме того, белки несопоставимо более разнообразны по составу и химическим свойствам, нежели нуклеиновые кислоты, поэтому, по мнению авторов статьи, на уровне белков можно запрограммировать гораздо больше функциональных возможностей, чем при подходе, предполагающем манипуляции с CRISPR. Возможно, использование белков в таком качестве позволит создавать даже совершенно новые клетки и ткани с заданными свойствами. Но белки, тем не менее, принципиально проигрывают ДНК по показателям прочности, репликации и долговечности, поэтому целесообразно их использовать для программирования клеток – например, как раз такой искусственной ткани или синтетических бактерий, которые предназначались бы именно для хранения и обработки информации.
Клеточный процессор
Наиболее сложной готовой разработкой в этой области представляется настоящий двухъядерный клеточный процессор, собранный в Высшей Технической школе Цюриха (на факультете биотехнологии и биоинженерии, расположенном в Базеле) в 2019 году. Этот процессор, работающий на основе технологии CRISPR, получен путем внедрения в живую клетку второго клеточного ядра. В качестве ввода он принимает молекулы РНК, регулируя экспрессию заданных генов и управляя таким образом синтезом белков. Исследователи подчеркивают, что в качестве такого процессора может работать единственная двухъядерная клетка, но ничто не мешает масштабировать его до целой ткани, содержащей миллиарды двухъядерных клеток. Теоретически такая система может превзойти по мощности современный суперкомпьютер, но потреблять лишь малую толику энергии по сравнению с таким суперкомпьютером. К тому же, на практике ничто не мешает внедрить в клетку не два, а сколько угодно ядер.
Клеточный компьютер может использоваться для детектирования биомаркеров в качестве ввода и для соответствующего реагирования на этот ввод. В качестве вывода клеточный компьютер может синтезировать молекулу лекарства, либо диагностического вещества. Подобное устройство может применяться, например, при лечении рака.
Здесь также нельзя обойтись без упоминания потрясающих возможностей биохимических компьютеров в областях, сближающихся с 3D-печатью, конструированием мемристоров и решением задач на поиск кратчайшего пути. На Хабре есть интересная статья «Нейросеть с амебой решили задачу коммивояжера», но амебы не сравнятся в поиске пути со слизевиками, которые способны захватывать жизненное пространство, вырастая от
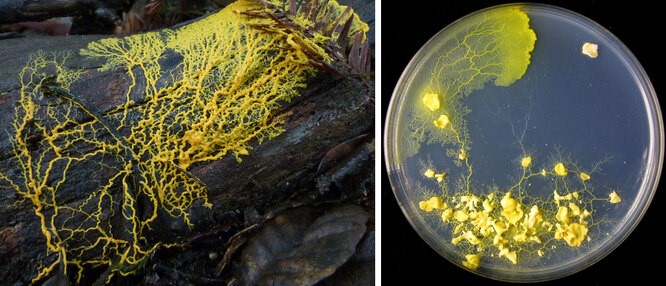
до

не обладая никакой нервной системой. Фактически, эти организмы состоят из сплошной цитоплазмы и захватывают окружающее пространство, достраивая себя в соответствии с некими топологическими алгоритмами. Например, слизевик успешно «запоминает», как прорасти к источнику пищи, даже если после достижения пищи отсечь его отросток. Программная реализация таких способностей позволила бы моделировать и создавать материалы с заданными свойствами, поскольку могла бы работать на клеточном и даже молекулярном уровне.
Здесь весьма кратко обрисованы возможности и проблемы биотехнологического программирования, но тема представляется очень перспективной во многом потому, что одни и те же законы клеточной репликации позволяют работать и с нуклеиновыми кислотами, и с белками/ферментами. Освоение подобных возможностей позволило бы сочетать долговечность и информационную избыточность ДНК (хранилище данных) с разнообразием белков (структуры данных), и не только кардинально решить проблему исчерпания возможностей закона Мура, но и полностью переосмыслить вычислительную технику, заменив software+hardware на wetware.

