
В контексте возраста планеты Земля человечество как вид существует не так давно. Однако за это время мы успели пройти невероятный эволюционный путь, в ходе которого нам открывались все новые и новые знания. С открытием письменности мы получили возможность фиксировать ту или иную информацию, дабы она была доступна другим спустя время. Технологический прогресс привел к цифровизации мира и формированию информационной сферы Земли, которая по некоторым оценкам насчитывает порядка 64 зеттабайт данных, т. е. Около 7 ×1013 Гб. Вполне ожидаемо, что вопрос хранения постоянно растущего объема данных стоит для современного общества весьма остро. Потому поиски новых методов хранения и новых типов хранилищ крайне важен. Ученые из Технического университета Эйндховена (Нидерланды) разработали новую методику хранения данных с помощью ДНК. В чем секрет данной методики, насколько работоспособны полученные хранилища, и сколько данных можно на них хранить? Ответы на эти вопросы мы найдем в докладе ученых.
Основа исследования
Проблема современного мира в аспекте информации заключается в том, что мы генерируем больше данных, чем можем хранить. Классические методы хранения (жесткие диски, магнитные ленты и т.д.) хоть и широко распространены, но обладают ограниченным сроком службы и, что важнее, ограниченной плотностью хранения. В связи с этим все больший интерес вызывают малые органические молекулы, полимеры и даже ДНК, как потенциальные хранилища данных.
Присущая ей способность к хранению информации, долговечность и высокая плотность информации делают ДНК главным кандидатом для хранения архивных цифровых данных. Заметный прогресс был достигнут в схемах кодирования, используемых для преобразования двоичных данных в ДНК, и лучший на сегодняшний день метод позволяет достичь плотности 17 экзабайт на грамм (около 1.8 х 1010 Гб), что примерно на шесть порядков превышает магнитные и оптические аналоги. Кроме того, длительное хранение ДНК в специальных оболочках из природных или синтетических материалов позволяет хранить данные намного дольше, чем это возможно в случае обычных магнитных носителей данных.
С появлением параллельного химического и ферментативного синтеза стало возможным крупномасштабное производство ДНК для хранения данных. В то же время секвенирование ДНК с использованием Illumina или метода на основе нанопор позволило достичь высокопроизводительного считывания последовательностей ДНК.
Обходя необходимость секвенировать целые наборы данных, закодированные в ДНК, произвольный доступ на основе полимеразной цепной реакции (ПЦР) может выборочно извлекать закодированные данные из сложного пула ДНК файлов. Однако у ПЦР есть два недостатка. Во-первых, небольшая часть пула безвозвратно расходуется во время амплификации данных и повторного копирования искаженной последовательности ДНК. Во-вторых, мультиплексный поиск файлов, закодированных ДНК, с использованием ПЦР затруднен из-за систематической ошибки ПЦР и образования артефактов из-за молекулярных перекрестных помех. Смещение ПЦР возникает из-за различий в длине, составе последовательности, содержании гуанина-цитозина и вторичной структуре ДНК.
Смягчить последствия образования артефактов и систематической ошибки можно путем тщательного проектирования последовательности и включения дополнительных физических и логических избыточностей, но это приводит к увеличению стоимости синтеза и секвенирования ДНК. Потому современные стратегии извлечения нескольких ДНК файлов основаны на физическом разделении реакций и индивидуальной амплификации каждого файла с использованием нескольких одиночных ПЦР реакций. Параллельная амплификация в одном реакционном сосуде может быть достигнута с помощью эмульсионной ПЦР, которая разделяет матрицы ДНК с помощью капель воды в масле и предотвращает образование артефактов. Хотя эмульсионная ПЦР использовалась для извлечения данных ДНК, сложный рабочий процесс, невозможность повторного использования и большое количество органических растворителей, необходимых для образования эмульсий для каждой реакции, делают ее малополезной для крупномасштабных систем.
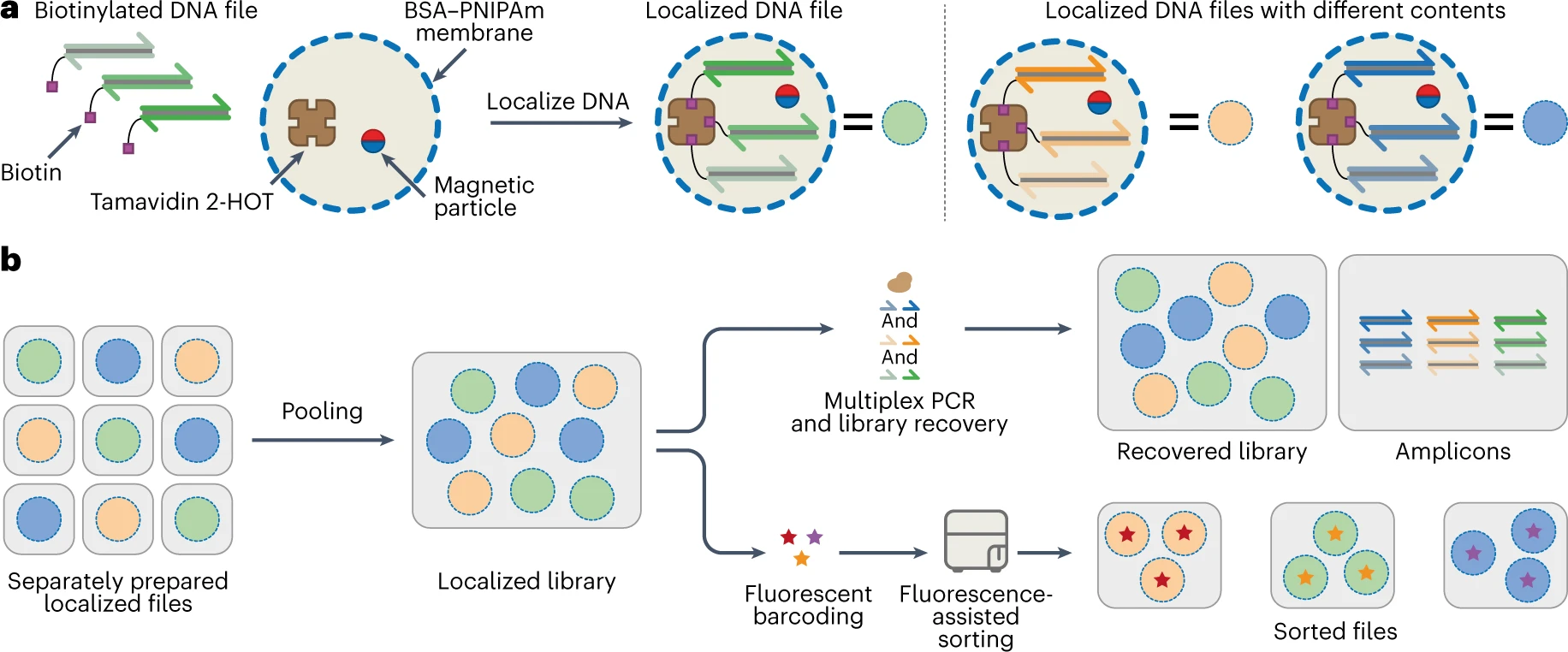
Изображение №1
В рассматриваемом нами сегодня труде ученые описывают метод, называемый «термоограниченной ПЦР», в котором используются микрореакторы с температурно-зависимой проницаемостью мембраны для повышения точности амплификации ПЦР. Данный метод позволяет реализовать мультиплексный, повторяющийся доступ на основе ПЦР к нескольким ДНК файлам из сложного пула ДНК. Метод основан на стабильной инкапсуляции файлов биотинилированной ДНК в отдельные популяции термочувствительных полупроницаемых микрокапсул (1a) с последующим объединением этих популяций.
Уникальная термореактивная проницаемость мембраны значительно снижает молекулярные перекрестные помехи и сопутствующее образование артефактов при температурах ПЦР, тем самым позволяя точно амплифицировать несколько ДНК файлов с закодированными данными, что сравнимо с эмульсионной ПЦР. Однако, в отличие от эмульсионной ПЦР, небиотинилированные ампликоны могут быть извлечены и отделены от исходных ДНК файлов, закодированных данными, при комнатной температуре без разрушения микрокомпартмента, поскольку проницаемость мембраны восстанавливается при комнатной температуре (1b).
В термоограниченной ПЦР используются протеиносомы — полупроницаемые микрокомпартменты на основе белково-полимерных конъюгатов («соединителей»). Биотинилированные ДНК-файлы могут быть стабильно локализованы внутри просвета протеиносом с помощью инкапсулированного биотин-связывающего белка тамавидин 2-HOT (1a).
Результаты исследования
Ранее учеными было установлено, что стрептавидинсодержащие протеиносомы могут быть получены путем ковалентного сшивания конъюгатов бычьего сывороточного альбумина (BSA от bovine serum albumin) и поли(N-изопропилакриламида) (PNIPAm) на границе раздела капель эмульсии вода-в-масле. Сшитые протеиносомы были фазово перенесены в воду, а добавление нитей, функционализированных биотином, или нитей двухцепочечной ДНК приводило к накоплению матрицы ДНК и стабильному удержанию до 200 пар оснований* (bp от base pair).
Спаренные основания* — пара двух азотистых оснований нуклеотидов на комплементарных цепочках нуклеиновых кислот, соединенная с помощью водородных связей.
Чтобы понять, как их термические свойства влияют на ПЦР инкапсулированных ДНК файлов, ученые исследовали температурно-зависимую стабильность и проницаемость протеиносом. Поскольку стрептавидин лишь частично устойчив к высоким температурам, используемым во время ПЦР, ученые использовали термостабильный аналог стрептавидина (тамавидин 2-HOT), который связывает биотин с аффинностью, аналогичной стрептавидину, но может выдерживать более высокие температуры во время ПЦР.
Были подготовлены протеиносомы, содержащие либо 4 мкМ стрептавидина, либо 4 мкМ тамавидина 2-HOT, чтобы проверить термостабильность обоих белков. Было подтверждено гомогенное распределение тамавидина 2-HOT внутри просвета. Также было установлено, что размер протеиносом, содержащих тамавидин 2-HOT, составляет 57 ± 17 мкм.

Изображение №2
Затем 188-нуклеотидную биотинилированную двухцепочечную ДНК A1F1, меченную Cy5, локализовали внутри подготовленных протеиносом путем инкубации их с двухцепочечной ДНК. Конфокальные снимки протеиносом показали, что после нагревания до 95 °C только протеиносомы, приготовленные с тамавидином 2-HOT, сохраняют встроенную двухцепочечную ДНК (2a). Для упрощения процесса извлечения протеиносом, в них также были включены суперпарамагнитные частицы. Вместе эти два изменения генерируют протеиносомы, содержащие термостабильную локализованную ДНК в сочетании с магнитным извлечением.
Поскольку PNIPAm становится частично несмешиваемым при температуре выше его более низкой критической температуры раствора (НКТР или LCST от lower critical solution temperature; ~ 32 °C), можно сделать вывод, что снижение проницаемости мембраны выше LCST может удерживать молекулы ДНК, полученные во время ПЦР-обработки захваченных ДНК файлов (2b). Предыдущее исследование показало, что нагревание протеиносом выше LCST снижает проницаемость мембраны для гидрофобных молекул, возможно, за счет увеличения гидрофобности мембраны и уменьшения размера пор.
Чтобы исследовать проницаемость одноцепочечной ДНК в зависимости от температуры, ученые сначала проверили, что протеиносомы, содержащие инкапсулированный тамавидин 2-HOT, проницаемы для одноцепочечной ДНК при комнатной температуре. Используя ранее разработанный массив микрожидкостных улавливателей, ученые захватили протеиносомы, содержащие тамавидин 2-HOT, и добавили в камеру улавливания 50-нуклеотидную одноцепочечную ДНК (оцДНК), меченную Alexa-546 (F2). Как и ожидалось, оцДНК легко диффундировала через мембрану. Поскольку оцДНК F2 не биотинилирована, промывание захваченных протеиносом приводит к быстрой потере флуоресценции. Это указывает на то, что мембрана протеиносомы обладает высокой проницаемостью для оцДНК при комнатной температуре (2c).
Затем ученые приготовили протеиносомы тамавидин 2-HOT, содержащие биотинилированную цепь A2 длиной 21 нуклеотид, спаренную с 10-нуклеотидным участком 50-нуклеотидной оцДНК F2, меченной Alexa-546 (прогнозируемая температура плавления, Tm = 65 °C), и нагрели протеиносомы до 95 °C, чтобы убедиться, что ДНК дуплекс полностью расплавился. Эксперименты с конфокальной визуализацией при этой температуре показали, что локализованная флуоресценция медленно уменьшалась с течением времени (2d). Этот результат, который можно объяснить либо небольшим количеством меченой Alexa-546 ДНК, диффундирующей через мембрану, либо артефактами визуализации, указывает на то, что проницаемость мембраны для относительно длинной одноцепочечной ДНК намного ниже при этой температуре.
В аналогичном эксперименте при 95 ° C с использованием более короткой оцДНК длиной 31 нуклеотид, меченной Alexa-546, ДНК быстро диффундировала через мембрану. После охлаждения до температур ниже LCST мембрана снова стала высокопроницаемой как для длинной, так и для короткой оцДНК. Это позволяет предположить, что изменение проницаемости мембраны, вызванное температурой, обратимо.
В совокупности эти результаты показывают, что протеиносомы тамавидина 2-HOT стабильно локализуют биотинилированную двухцепочечную ДНК при более высоких температурах даже после расплавления ДНК дуплекса и что высокая проницаемость мембраны восстанавливается при охлаждении до температур ниже LCST.
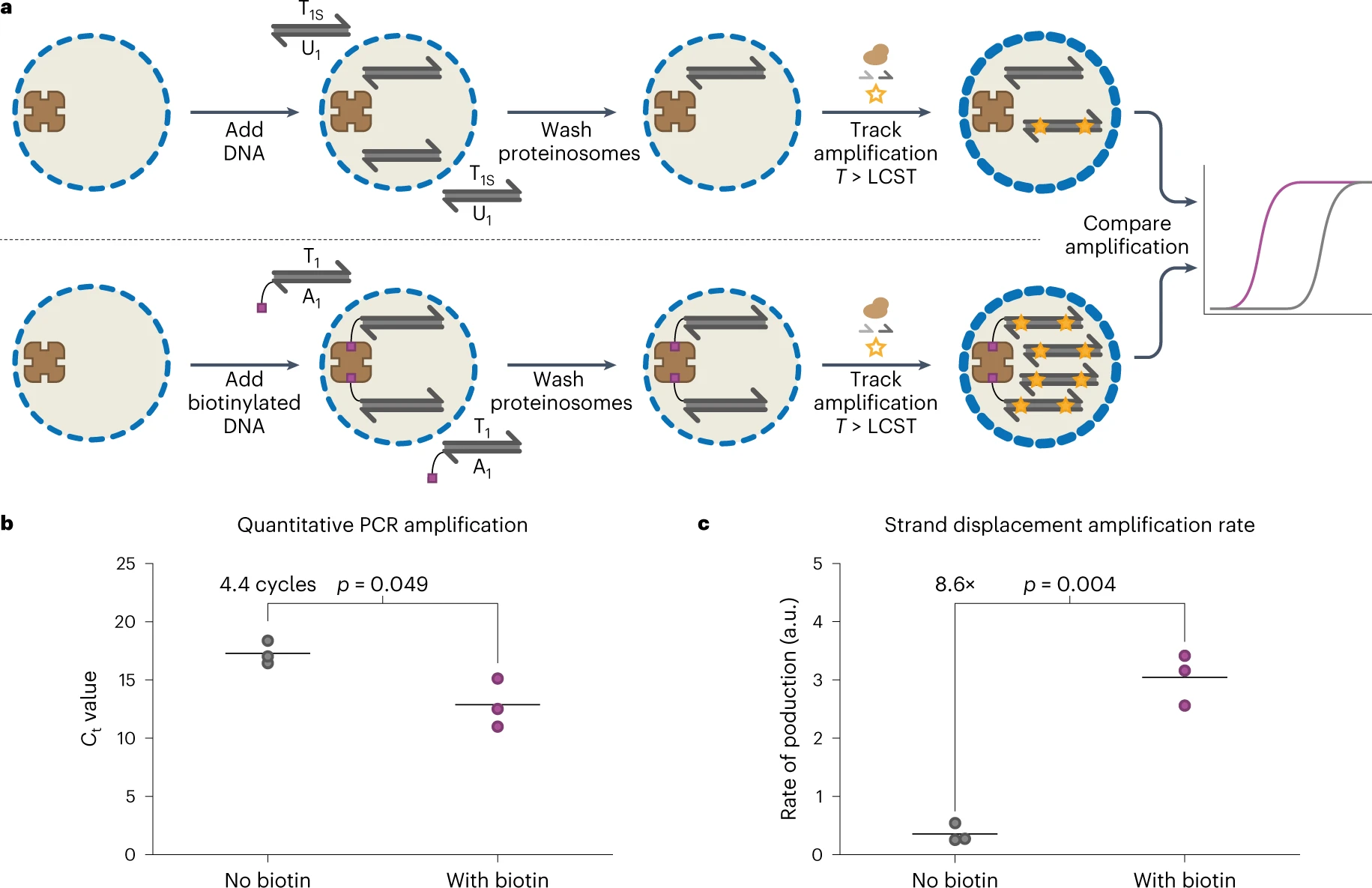
Изображение №3
Далее ученые приступили к ферментативной амплификации локализованных ДНК матриц. Ранее для амплификации молекул ДНК, кодирующих данные, использовались как ПЦР, так и амплификация со смещением цепи (SDA от strand displacement amplification).
Чтобы продемонстрировать общую применимость протеиносом, содержащих встроенный тамавидин 2-HOT, для поиска ДНК данных, ученые показали, что локализованная биотинилированная ДНК с длиной, обычно используемой для хранения данных, может быть амплифицирована с использованием ПЦР или SDA (3a).
Чтобы измерить локализованную амплификацию двухцепочечной ДНК с помощью ПЦР in situ (в естественной среде), была использована количественная ПЦР (кПЦР). Протеиносомы, содержащие 4 мкМ тамавидина 2-HOT, инкубировали с 300 нМ биотинилированной 178-нуклеотидной матрицы двухцепочечной ДНК T1A1 либо небиотинилированной 168-нуклеотидной контрольной двухцепочечной ДНК T1SU1.
После промывки для удаления свободной матрицы была добавлена реакционная смесь для ПЦР, состоящая из полимеразы, дезоксинуклеозидтрифосфатов (дНТФ), праймеров и чувствительного к двухцепочечной ДНК флуоресцентного красителя EvaGreen. Для каждой реакции определяли пороговый цикл для оценки количества ДНК, доступного для амплификации. В среднем для достижения порога флуоресценции требовалось на 4.4 цикла меньше, когда вместо небиотинилированной двухцепочечной ДНК использовали биотинилированную двухцепочечную ДНК (3b). Предполагая идеальную амплификацию во время каждого цикла ПЦР, это указывает на то, что примерно в 21 раз больше двухцепочечной ДНК доступно для амплификации, когда двухцепочечная ДНК биотинилирована.
Затем была использована SDA для изотермической амплификации локализованной двухцепочечной ДНК. Протеиносомы, содержащие 4 мкМ тамавидина 2-HOT, инкубировали с 300 нМ биотинилированной матричной двухцепочечной ДНК T1A1 или немеченой T1SU1 перед промывкой и последующим добавлением смеси для амплификации, состоящей из полимеразы, одноцепочечного ДНК-связывающего белка, никазы, дНТФ, праймеров и EvaGreen.
SDA реакции протеиносом, содержащих T1A1, показали в среднем в 8.6 раза более высокую скорость амплификации, чем протеиносомы, инкубированные с небиотинилированным T1SU1 (3c).
В совокупности вышеописанные результаты, полученные с помощью ПЦР и SDA, показывают, что биотинилированная двухцепочечная ДНК, локализованная с помощью тамавидина 2-HOT и имеющая длину, обычно используемую для хранения данных ДНК, может быть амплифицирована изнутри протеиносом.
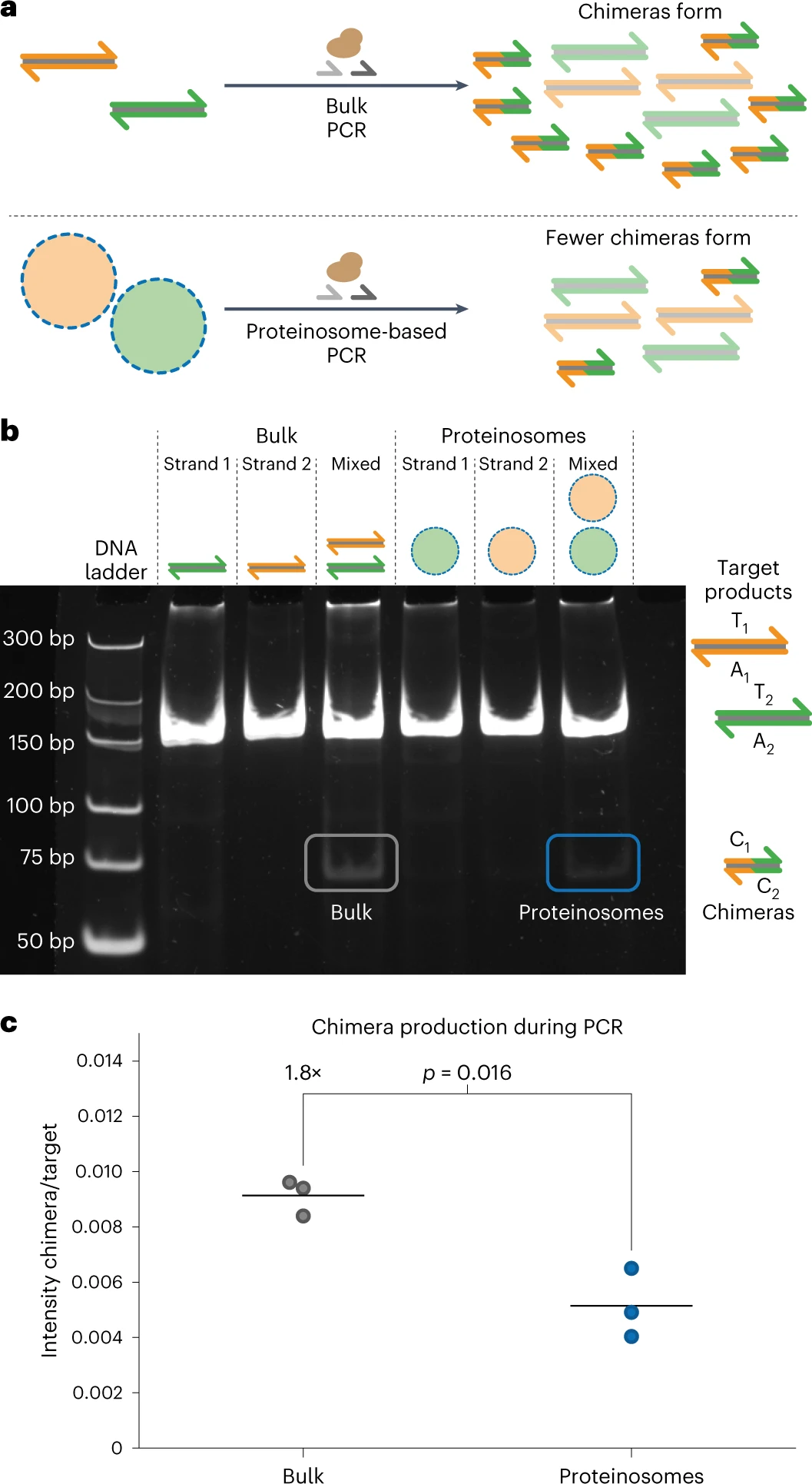
Изображение №4
Произвольный доступ на основе ПЦР может извлекать закодированные данные из сложных пулов ДНК. Однако последовательности, содержащие очень похожие короткие участки, подвержены рекомбинации во время ПЦР. Формирование таких химерных ампликонов портит ДНК файлы, поскольку данные вставляются в неверные позиции. Кроме того, образование химер приводит к систематической ошибке ПЦР в сложных пулах ДНК, которая ранее ингибировалась с помощью эмульсионной ПЦР. Терморегулируемая проницаемость мембран протеинсом должна снижать образование химер во время мультиплексной ПЦР сложных пулов ДНК, поскольку матрицы эффективно разделяются при типичных температурах ПЦР (4a).
Чтобы исследовать, как термоограниченная мультиплексная ПЦР из матриц, локализованных в протеиносомах, влияет на формирование химеры, были разработаны два набора биотинилированных матриц двухцепочечной ДНК длиной 178 нуклеотидов (T1A1 и T2A3), которые имеют общую комплементарную область из 31 нуклеотида, так что химера C1C2, образованная во время мультиплексной ПЦР, имеет длину 71 bp (4b).
Чтобы локализовать две матрицы внутри отдельных протеиносом, содержащих 4 мкМ тамавидина 2-HOT, было добавлено 300 нМ двухцепочечной ДНК, а затем выполнено промывание микрокапсулы для удаления избытка ДНК. Популяции протеиносом смешивали и амплифицировали с помощью мультиплексной ПЦР, а ампликоны выделяли при комнатной температуре.
В качестве контрольного эксперимента матрицы также амплифицировали в массовых реакциях. Анализ с помощью электрофореза в полиакриламидном геле (PAGE от polyacrylamide gel electrophoresis; 4b) выявил образование химерного продукта, а именно C1C2 длиной 71 bp в общей реакции.
Термокоограниченная мультиплексная ПЦР также показала слабую полосу на уровне 71 bp, но эта полоса имеет значительно более низкую интенсивность по сравнению с объемной ПЦР. Обе реакции продуцировали примерно одинаковое количество ДНК-мишени, судя по интенсивности полос 178 bp. Чтобы количественно оценить эффект термоудержания во время мультиплексной ПЦР, ученые использовали Fiji для измерения интенсивности целевых и химерных полос.
Ученые использовали интенсивность целевой полосы в качестве внутреннего контроля для учета изменчивости от образца к образцу и рассчитали отношение химеры к продукту для каждой реакции (4c). Объемные реакции давали более высокие отношения химер к мишеням, указывая на то, что значительно больше химер образуется в объемных условиях по сравнению с локализованной в протеиносомах амплификации. Некоторое образование химерной ДНК наблюдалось при мультиплексной ПЦР с использованием протеиносом, что можно объяснить неполным удалением нелокализованной ДНК или ограниченным высвобождением ампликонов из протеиносом.
Несмотря на то, что полной супрессии химер, как и ожидалось для эмульсионной ПЦР, не наблюдалось, этот результат продемонстрировал, что термоограниченная мультиплексная ПЦР в протеиносомах значительно снижает образование химер за счет локализации амплификации ДНК в отдельных компартментах.

Изображение №5
Показав, что пространственное разделение реакций ПЦР снижает образование химер, ученые пришли к выводу, что локализация файлов, закодированных данными, внутри протеиносом Тамавидин 2-HOT должна лучше поддерживать представление файлов, поскольку образование химер отрицательно влияет на систематическую ошибку ПЦР.
Чтобы продемонстрировать это, двадцать пять файлов размером 1 МБ, в общей сложности 25 МБ, были закодированы в последовательности ДНК из 110 оснований с использованием ранее описанного метода. Каждый файл, состоящий примерно из 66000 уникальных последовательностей, был локализован внутри отдельных популяций протеиносом Тамавидин 2-HOT. Затем 25 популяций объединяли, чтобы создать библиотеку на основе протеиносом. Затем файлы были амплифицированы с помощью мультиплексной ПЦР в объемном растворе или в разделенных средах, содержащих либо капли эмульсии вода-в-масле, либо популяции протеиносом в воде. Относительные концентрации файлов после мультиплексной ПЦР были количественно определены с использованием количественной ПЦР для определения доли каждого файла.
Данные количественной ПЦР показали, что термоограниченная и эмульсионная ПЦР сохраняют распределение файлов более эффективно, чем массовая амплификация. Затем было выполнено секвенирование Illumina (5a) для определения среднего покрытия на файл, чтобы проверить, переводится ли улучшенная пропорциональность в более однородное и равномерное покрытие на файл. Среднее покрытие на файл, нормализованное к среднему значению, было построено в логарифмическом масштабе, чтобы показать отклонение покрытия в порядках величины (5b).
Как и ожидалось, наблюдалась большая 60-кратная разница в покрытии между наиболее и наименее представленными файлами, амплифицированными в массовых условиях. Напротив, файлы, амплифицированные с использованием локализованных реакций, показали гораздо меньшие разбросы семи- и пятикратных различий для ПЦР на основе протеиносом и эмульсии соответственно. Начальный разброс, присутствующий в библиотеке до амплификации, был трехкратным.
Кроме того, были определены коэффициенты вариации (CV от coefficient of variation) для всех состояний в качестве меры равномерности распределения. CV исходного пула, объемно-амплифицированной ДНК, капель эмульсии и протеиносом составляли 24%, 139%, 35% и 52% соответственно. Эти результаты показывают, что термоограниченная ПЦР является работоспособной альтернативой эмульсиям на масляной основе для пропорциональной мультиплексной амплификации данных, закодированных в ДНК.
Поскольку биотинилированная ДНК остается локализованной внутри протеиносом Тамавидин 2-HOT, ожидается, что восстановление протеиносом, а также их инкапсулированных ДНК-кодированных файлов с использованием магнитной сепарации после ПЦР обеспечивает надежный повторный доступ к данным, закодированным в ДНК. Чтобы проверить это, три файла были локализованы внутри протеиносом, содержащих 4 мкМ тамавидина 2-HOT. Перед объединением протеиносомы промывали для создания библиотеки на основе протеиносом. Кроме того, файлы также были смешаны в эквимолярных концентрациях в массе для создания нелокализованной библиотеки. Из полученных библиотек файлы амплифицировали в трех последовательных раундах объемной, эмульсионной или протеиносомной ПЦР, каждый раунд извлекая другой файл. После трех раундов был снова получен доступ к исходному файлу.
Библиотеку, амплифицированную с помощью объемной ПЦР, очищали путем инактивации dNTP и праймеров перед использованием для амплификации следующего файла. Наконец, библиотеку, амплифицированную с помощью эмульсионной ПЦР, очищали путем разрушения эмульсии с последующей очисткой на спин-колонке.
Используя секвенирование Illumina, ученые определили выпадение в файле, т.е. долю последовательностей, которые должны быть частью файла, но больше не наблюдаются (5c). Было установлено, что повторный доступ к ПЦР библиотеке на основе протеиносом привел к наименьшей потере последовательностей, за которой следовали эмульсионная ПЦР и объемная ПЦР (5d).
Как говорят ученые, получение файлов ДНК с помощью ПЦР очень специфично, однако потребность в достаточно ортогональных праймерах ограничивает количество файлов, которые можно хранить в одном пуле. Это ограничение привело к разработке дополнительных стратегий случайного доступа к файлам ДНК, таких как физическое разделение, поиск на основе гибридизации, альтернативные схемы амплификации и сортировка с помощью флуоресценции. В дополнение к демонстрации многократной мультиплексной ПЦР ученые стремились разработать методы выборочного извлечения файлов из библиотек протеиносом, кодируемых в ДНК.

Изображение №6
Для достижения этой цели была разработана стратегия флуоресцентного штрих-кодирования популяций протеиносом, основанная на двух процессах: мечение мембраны микрокапсул изотиоцианатом флуоресцеина (FITC) или DyLight 405; добавление коротких биотинилированных оцДНК, меченных Cy3 (F4) или Cy5 (F5), после того, как файлы исходной кодированной ДНК были локализованы внутри протеиносом (6a).
Этот подход позволяет различать четыре популяции протеиносом, хотя теоретически возможно генерировать до 2N уникальных комбинаций штрих-кодов, где N — количество флуорофоров, используемых для штрих-кодирования.
После инкапсуляции файлов, кодирующих данные, в протеиносомы со штрих-кодом, отдельные популяции объединялись, затем использовалась сортировка клеток с активацией флуоресценции (FACS от fluorescence-activated cell sorting) для выбора конкретных файлов из пула (6b) с помощью трехэтапной процедуры отбора.
Во-первых, протеиносомы были отобраны на фоне невключенных магнитных частиц и фона с использованием стробирования на каналах FSC-A и FSC-H.
Во-вторых, мембранные метки FITC и DyLight 405 использовались в качестве флуоресцентных границ, поскольку присутствие или отсутствие флуорофоров приводило к четким бимодальным распределениям в обоих каналах (слева на 6c).
В-третьих, были установлены флуоресцентные границы Cy3 и Cy5, чтобы различать локализованные субпопуляции F4 и F5 (справа на 6c).
Затем объединенные протеиносомы были отсортированы на четыре разные популяции на основе их различных флуоресцентных характеристик: высокий уровень DyLight 405 / высокий уровень Cy5; высокий DyLight 405 / высокий Cy3; высокий FITC / высокий Cy3; высокий FITC / высокий Cy5.
Отсортировав пул на отдельные популяции, была использована количественная ПЦР для определения долей каждого файла в разделенных популяциях, чтобы подтвердить, что были получены правильные данные, закодированные в ДНК (6d). Было обнаружено, что предполагаемые файлы составляют в среднем 75.0% всей ДНК в отсортированных образцах.
Для архивного хранения данных на основе ДНК долгосрочная стабильность ДНК имеет решающее значение. Предыдущая работа показала, что лиофилизация ДНК увеличивает гидролитическую стабильность ДНК примерно на порядок. Ученые провели лиофилизацию протеиносом, содержащих встроенную ДНК, в присутствии трегалозы — лиопротектора, который дополнительно повышает стабильность ДНК. Эксперименты с микроскопией и количественной ПЦР показали, что регидратация твердого порошка не влияла на целостность ДНК и совместную локализацию внутри протеиносом. Слияния протеиносом после регидратации также не наблюдалось.
Для более детального ознакомления с нюансами исследования рекомендую заглянуть в доклад ученых и дополнительные материалы к нему.
Эпилог
Достижения в области синтеза и секвенирования ДНК сделали возможным долгосрочное хранение цифровых данных о ДНК. Однако повторный и параллельный произвольный доступ на основе ПЦР до сих пор был проблематичным. В рассмотренном нами сегодня труде ученые успешно разработали новый метод, основанный на термочувствительных полупроницаемых микрокомпартментах, который обеспечивает произвольный доступ на основе мультиплексной ПЦР с характеристиками, сравнимыми с эмульсионной ПЦР. Однако, в отличие от эмульсионной ПЦР, инкапсулированные ДНК-кодированные файлы остаются локализованными внутри микрокомпартментов после амплификации и извлечения данных, что позволяет повторно копировать исходные молекулы, кодирующие файл. Кроме того, ученые включили штрих-коды на основе флуоресценции и использовали их для сортировки файлов на основе протеиносом, обеспечивая дополнительный уровень организации данных.
Основными преимуществами новой методики является относительно небольшая стоимость и возможность получить повторный доступ к закодированным в ДНК файлам, несмотря на более длительный процесс кодирования. В будущем ученые намерены провести ряд исследований для улучшения стабильности инкапсулированной ДНК в высушенном состоянии с использованием методов ускоренного старения. Так они хотят узнать, как можно продлить срок службы таких необычных хранилищ данных.
Немного рекламы
Спасибо, что остаётесь с нами. Вам нравятся наши статьи? Хотите видеть больше интересных материалов? Поддержите нас, оформив заказ или порекомендовав знакомым, облачные VPS для разработчиков от $4.99, уникальный аналог entry-level серверов, который был придуман нами для Вас: Вся правда о VPS (KVM) E5-2697 v3 (6 Cores) 10GB DDR4 480GB SSD 1Gbps от $19 или как правильно делить сервер? (доступны варианты с RAID1 и RAID10, до 24 ядер и до 40GB DDR4).
Dell R730xd в 2 раза дешевле в дата-центре Maincubes Tier IV в Амстердаме? Только у нас 2 х Intel TetraDeca-Core Xeon 2x E5-2697v3 2.6GHz 14C 64GB DDR4 4x960GB SSD 1Gbps 100 ТВ от $199 в Нидерландах! Dell R420 — 2x E5-2430 2.2Ghz 6C 128GB DDR3 2x960GB SSD 1Gbps 100TB — от $99! Читайте о том Как построить инфраструктуру корп. класса c применением серверов Dell R730xd Е5-2650 v4 стоимостью 9000 евро за копейки?


