
Три года назад в Artezio в поисках партнера для разработки софта обратилась британская компания, занимающаяся автоматизацией анализа и интерпретацией генетических исследований. Основная миссия компании – это внедрение генетической медицины в стандартный процесс здравоохранения, создание системы поддержки принятия клинических решений. Найти значимые генетические аномалии в задумке должно быть не сложнее, чем сдать анализ крови. Сейчас с нашим back-end разработчиком Артемом расскажем, как работают такие проекты.
Roots: от исследований к практике
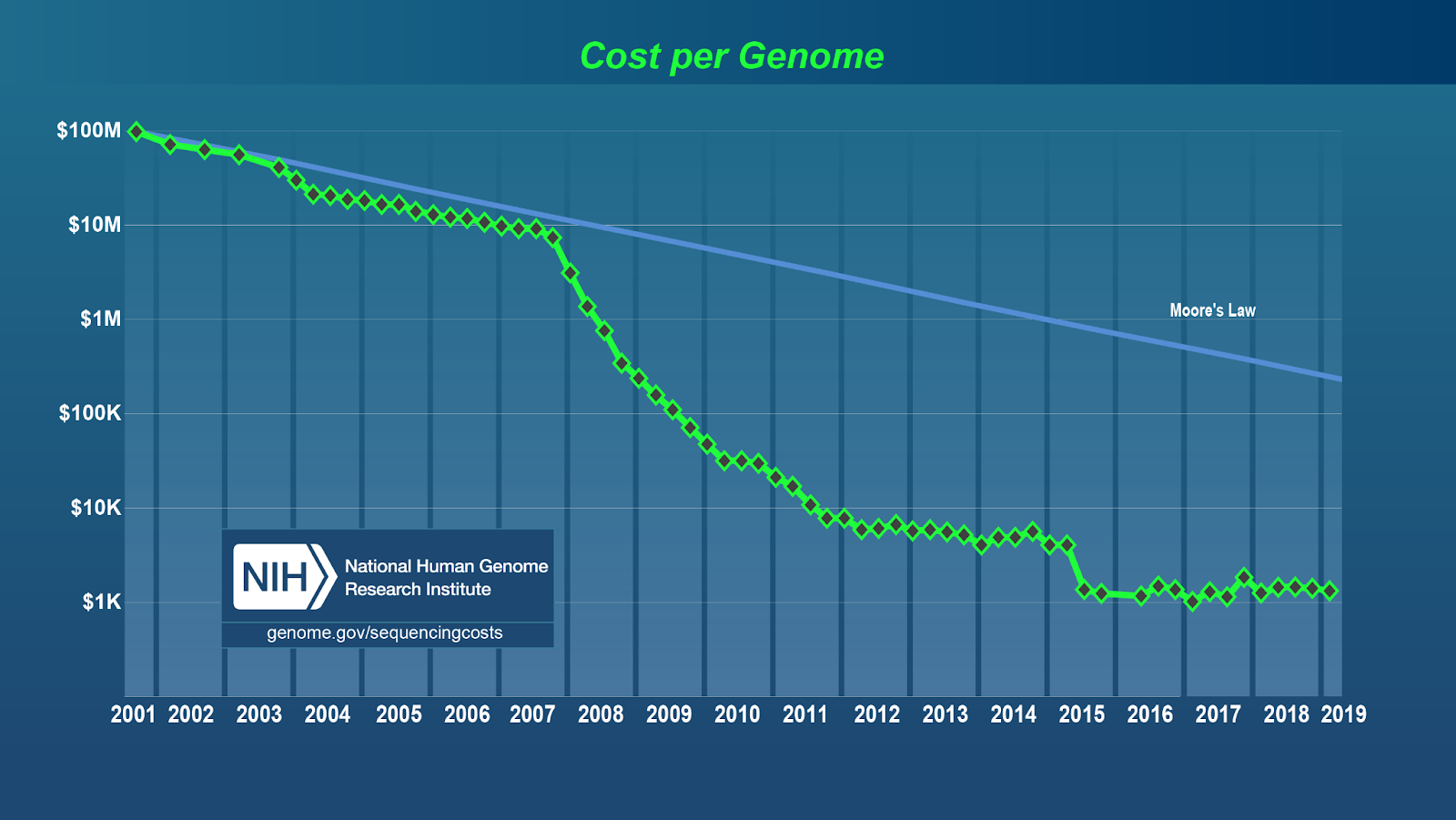
Последние двадцать лет стоимость генетических исследований падала экспоненциально, и это позволило всему миру перейти от научных исследований к регулярной медицинской практике в этой области.
Англия очень правильное место, для того чтобы заниматься генетическими исследованиями. В середине прошлого века там открыли структуру ДНК, за что впоследствии Уотсон, Крик и Уилкинс удостоились Нобелевской премии. Британия всегда была на передовой геномных исследований, собственно и основатели компании-заказчика – ученые-генетики, руководители крупных научных и государственных проектов, а в основе платформы – технологии, разработанные в институте Сенгера.
Кому и зачем нужен геномный анализ
Клиентами нашего заказчика являются клинические лаборатории по всему миру. Но, вероятно, главный среди них — Национальная служба здравоохранения Великобритании. История сотрудничества с ней начинается с проекта «100000 Genomes» (его инициировала государственная компания Genomics England), есть и похожий китайский проект «100K Wellness Pioneer Project». В России, кстати, тоже есть подобный проект «100000 геномов», но пока о нем мало информации, а та, что доходит, довольно спорная.
Модель обслуживания SaaS — геномный анализ as a service. Вы — клиническая лаборатория, у вас есть оборудование, позволяющее методами секвенирования нового поколения получить информацию о генетических данных пациента. Дальше вам необходимо проанализировать эту информацию и понять, есть ли какие-то странности в геноме пациента, которые могут вызывать те или иные болезни.
Надо сказать, что генетическая информация хранится в весьма запутанном виде и совсем не всегда по ней можно получить однозначный ответ. Заболевание может вызвать совокупность нескольких мутаций, да еще и в сочетании с факторами внешней среды. Существует практически бесконечное количество вариантов нормы, как и масса вариантов отклонений, разобраться во всем этом непросто. С точки зрения данных, геном человека представляет из себя строку из трех миллиардов символов ‘C’, ’G’, ’A’ и ’T’, при этом активных генов примерно 1,5%, все остальное – некодирующая “мусорная” ДНК. И в каждом геноме, как правило, есть и пропущенные буквы, и вставленные лишние, и случайные замены. В силу объема информации искать в геноме (или экзоме) причину заболевания — это как искать иголку в стоге сена. Но если искать сотни тысяч иголок в сотне тысяч стогов, то оказывается, что можно существенно ускорить этот процесс.
Основное направление деятельности нашего заказчика — редкие и наследственные генетические заболевания (сейчас компания также активно развивает анализ данных в области персональной медицины в онкологии, ведении беременности и борьбе с психическими заболеваниями). Считается, что существует 7–8 тысяч редких и наследственных заболеваний, 80% из которых связаны с генетическими отклонениями. Сюда входят самые разные болезни: как пороки сердца, глухота, так и сложные мультисистемные заболевания, часть из них слабо исследована, найти причину отклонения и понять, как лучше помочь пациенту, не так-то просто.

Как это работает и при чем тут The Human Genome Project
Вот тут-то на помощь лаборатории приходит решение нашего заказчика. Лаборатория отправляет полученную в ходе исследования информацию в пайплайн (через API или человеческий интерфейс) и быстро получает в ответ результаты (хотя, может быть, и не всегда исчерпывающие). Существует условный референсный геном человека, собранный на основе результатов деятельности международного научно-исследовательского проекта «The Human Genome Project» в начале нулевых, существуют медицинские базы знаний различных генетических вариаций (например, ClinVar, DECIPHER – одни из крупнейших), также у каждой лаборатории могут быть свои подобные базы, у нашего заказчика, конечно, есть и свои. Вообще собрать все эти базы, поддерживать данные в актуальном состоянии – одна из очень непростых задач, т.к. базы часто разрознены, созданы небольшими группами ученых, устаревают, ПО, работающее с этими базами, не поддерживается. Собрать это все и интегрировать – одна из решаемых задач.
Далее алгоритмы (в том числе и ИИ) ищут значимые мутации по базам знаний генетических вариаций. Система находит встречающиеся отклонения и выдает отчет с результатами исследования. При этом конечное решение об интерпретации всегда принимает сотрудник лаборатории, но скорость анализа при помощи нашего решения существенно (утверждается, что в десять раз и более) увеличивается, а нагрузка на специалистов из лабораторий снижается. Фактически подключать специалистов требуется в действительно сложных и запутанных случаях, а интерпретацию типовых, т.е. большей части материала, — проводить в автоматическом режиме.
Время, затрачиваемое на интерпретацию результатов, сокращается, пропускная способность лаборатории увеличивается, т.е. удается поставить этот процесс на поток и встроить генетические исследования в систему здравоохранения как еще один стандартный инструмент, например, как анализ крови.
Если еще больше приблизиться к реальности, то вот пример из практики. Одиннадцатимесячная девочка госпитализирована в реанимацию с припадком неясного генеза. В лаборатории было достаточно быстро произведено секвенирование ее генетических данных, и затем они были загружены в автоматический пайплайн, который через 8 минут выдал результат интерпретации, подсветив отклонения в гене, связанном с аутосомной эпилепсией, о которой родители девочки не имели никакого понятия. Т.е. ответ о причинах заболевания был получен очень быстро, а это позволило оперативно принять решение о лечении и необходимой помощи, быстрее помочь пациенту и его родным. Скорость принятия решения в таких случаях может предотвратить такие опасные последствия, как необратимые повреждения головного мозга.
А теперь расскажем о том, как этот проект выглядит с нашей стороны, как IT-компании в целом и back-end разработчика в частности. Дальше передаю слово своему коллеге — back-end разработчику Артему.
Контакт с заказчиком, языковой барьер, культурные ценности, SCRUM, процессы, инициатива
На проект я попал через обычное интервью на инженера backend-разработки. Свободный разговорный английский — один из важнейших критериев для нашего заказчика. Так как заказчик соблюдает много scrum — и agile-ритуалов, сотрудникам нужно ежедневно присутствовать на встречах, высказывать свою точку зрения, обсуждать решения. Все это культивирует заказчик, и мы в Artezio подстраиваемся под эти процессы. Соответственно, при приёме на проект знание английского имеет вес наравне с техническими знаниями. Входное интервью было полностью на английском. Собеседовали инженеры заказчика, интервью было организовано ими.
Что касается культурных ценностей, то в команде заказчика присутствуют разные сотрудники, в том числе иммигранты из других стран, люди разных поколений, есть и еще иностранные вендоры кроме нас, так что к разнице менталитетов здесь все привыкли. К тому же у Artezio опыт работы с британскими компаниями из области здравоохранения начинается с 2005 года.
На проекте с десяток команд, все идут в одинаковом темпе двухнедельных спринтов, каждый второй четверг у всех команд закрывается спринт и открывается новый. Этот день полон on-line встреч, в том числе запускается демоверсия приложения и все команды показывают, что они сделали. У нас команда 404. Название придумывалось как временное, но слишком всем понравилось, чтобы менять. Разрабатываем новую часть системы, которая занимается онкологией. Мы анализируем клетки и белки опухолей.
У нас отличная команда. Бывают небольшие задержки, но решаются они быстро. Зависших тикетов на доступы нет. Решения, связанные с дизайном и архитектурой, принимаются очень просто — на звонке. Все договариваются, и итоговое решение реализуется очень быстро. Команда работает слаженно. Руководство тоже помогает: Project owners объясняют, например, биоинформатическую часть, в которой нам приходится разбираться в связи со спецификой проекта. Нам нужно понимать, какие белки что значат, какие показатели на что влияют и так далее. Сначала было сложно разобраться в обилии сокращений и аббревиатур, но у заказчика классная Wiki. В ней задокументировано вообще все. В том числе есть глоссарий сокращений из биоинформатики. Его можно сразу выучить и понимать, о чем идет речь на звонках и встречах.
От стартапа у заказчика сохранилась легкость в принятии решений. Здесь все можно решить звонком. Хотя видно, что сейчас это серьезная организация с качественно построенными процессами. Все общаются очень открыто, есть межкомандное взаимодействие, все знают, кто над чем работает. Процесс разработки построен грамотно, можно менять решения, применять новые технологии. Так вышло, например, с Pydantic. Его просто предложили в чате, а у меня был опыт работы с ним на прошлом проекте, и я занялся его интеграцией. Показал, что Pydantic может, а может он все. Об этом ниже. Это круто, когда твой прежний опыт приносит пользу в новых проектах. Словом, всегда есть место диалогу, и предложения «снизу» также котируются.

Технологии, стек
Моя работа — все, что связанно с backend на Python. Я же занимаюсь настройкой и разработкой окружений для Python-части проекта. У нас есть разделение: часть обработки данных находится на Python, часть — на Perl, следы изначальной архитектуры, которую иногда переделывают и заново создают уже на Python. Так что постепенно Python-часть увеличивается. Python-часть проще поддерживать, потому что Perl-специалистов не так много. На этом проекте активно используется Kubernetes. Раньше я с ним не сталкивался. В остальном набор технологий стандартен для проектов, которые сейчас существуют. Во Frontend’е — JavaScript, в автоматизации тестирования – Selenium и Python.
Мы занимаемся разработкой пайплайнов. Для меня это принципиально новое занятие. Пайплайн – это системы обработки данных на всех шагах. Тут можно поучаствовать в разработке архитектуры. И комбинация технологий здесь довольно удачная. Приходится работать с разными частями системы, не только с Python-кодом, но и с Docker-файлами, Kubernetes и настройкой окружения. Задачи получаются шире и интереснее. Иногда даже приходилось вникать в Perl-код. К счастью, у нас есть разработчик, к которому мы всегда обращаемся за помощью. Docker контейнеры, в которых выполняется код, собираются для более легкой загрузки на сервер и устанавливаются со всеми необходимыми приложениями. Это все оркестрируется в Kubernetes. Для API у нас используется Flask, для базы данных — PostgreSQL. В том числе используем Pydantic и Jenkins, которые мы настроили с нуля.
Мы занимаемся генотипированием образцов опухолей, и у нас есть много файлов с информацией о генах и хромосомах. Каждая строчка в файле соответствует специфическому участку хромосомы, и каждая колонка соответствует информации о данной участке, связанных с ним генах. Данные анализируются набором внутренних и сторонних библиотек, после чего с результатами нужно сделать ещё некоторые манипуляции и в итоге выдать один красивый JSON-объект. Возник вопрос, как это сделать более компактно.
Было решено использовать Pydantic-библиотеки (Python) для работы с данными, которые как раз уменьшают размер кода и требуют меньше трудозатрат. У нас получилось красивое решение: один инструмент для множества различных колонок, которые приводятся к единому виду в конце. То есть любой набор колонок и строк, имеющийся на входе, превращается в JSON-объект нужного формата, причем информация берется из разных файлов и транслируется в один. Довольно удобно.
Думаю, мне поможет в дальнейшем то, что здесь присутствует аспект перманентного обучения. Не всегда знаешь, как работают все технологии в стеке. В предыдущих командах были DevOps, которые занимались только Docker, поэтому у нас не было необходимости глубоко его знать. В данном случае мы почти каждый день создаем образы, разбираем их, пересобираем, меняем конфигурацию, приходится разбираться в Docker.

Защита персональных данных пациентов, требования к лабораторным исследованиям
Одно из ключевых требований проекта — соответствовать стандартам по обработке личных данных пациентов. Существует множество стандартов и требований, регулирующих работу в медицине на уровне законодательства стран. Эти стандарты и требования периодически меняются и дорабатываются. Есть американский стандарт – HIPAA, европейские стандарты, в том числе и по обработке личных данных любого человека без привязки к роли пациента — GDPR, регламент (EU) 2017/746 (IVDR) о медицинских изделиях для диагностики в лабораторных условиях. Чтобы обеспечить выполнение этих законов и гарантировать, что процессы обработки информации внутри ПО соответствуют всем правилам, нужно принимать дополнительные действия при тестировании и анализе кода. На нас, как на поставщиках, лежит часть этой ответственности, мы тоже должны соответствовать этим процессам и принимать все меры для соблюдения законодательства в сфере регулирования данных пациентов. Это важно. Для этого у нашего заказчика уже выработаны и описаны процессы и правила, которым мы должны следовать, подготовлены тренинги.
Улучшать мир и себя
Я думаю, большая удача, когда тебе удается поучаствовать в таком интересном и полезном для человечества проекте, ощутить себя причастным к прогрессу. На проекте проводится много презентаций для сотрудников. Обсуждается, например, что такое рак, откуда он взялся, что мы о нем знаем. Для слаженной работы на таких проектах важно понимать теоретическую часть прикладной сферы. Сейчас, работая с онкологическими данными, я чувствую, что это полезно, актуально, и к тому же в этом процессе присутствует элемент образования и самообразования.
Кстати, прямо сейчас мы ищем python разработчика и биоинформатика для этого проекта. Эти и другие (Java / Python / Mobile / QA) вакансии на нашем сайте.


