
Краткий экскурс в генетику. Если вдруг вы помните, в 2003 г. было сделано сенсационное заявление: ученые, наконец, расшифровали геном человека. Геном построен из ДНК, а ДНК – это исходный код организма. ДНК представляет собой двойную цепочку, состоящую из 4-х видов нуклеотидов, которые повторяются в геноме человека порядка 3 млрд. раз. Так же, как в битах зашифрована вся информация на вашем компьютере, в нуклеотидах зашифрована инструкция о сборке всех белков человеческого тела. То есть зная, в какой последовательности расположены нуклеотиды в ДНК, мы теоретически можем собрать все необходимые белки и получить модель человека. Так вот в стандартном понимании ученые не расшифровали ДНК, а просто перевели химическую последовательность в набор нулей и единиц на компьютере. Что делать с этим дальше – отдельный разговор. Например, на данный момент нам ясна функция лишь 5% всего массива генома (это кодирование белков). Чем занимаются остальные 95%, можно только предполагать.
В 2003 году стоимость секвенирования ДНК человека составляла около 100 млн долларов. С течением времени эта цифра уменьшалась и сейчас она приближается к тысяче долларов. Вы платите, вашу ДНК секвенируют и отдают вам жесткий диск с 3 ГБ информации – вашим геномом в цифровом виде.
Сегодня на рынке представлено три основных секвенатора. Самый производительный, Hiseq, и его приемник NovaSeq, обеспечивает самое дешевое (флуоресцентное) секвенирование. Один его запуск длится несколько дней, и за это время обрабатываются геномы сразу нескольких человек. Однако сам запуск стоит около десятка тысяч долларов. К слову, и сам прибор стоит порядка $1 млн, а, поскольку устаревает он примерно за 3 года, для того, чтобы он окупился, он должен приносить вам $1000 в день.
Второй прибор появился на рынке буквально прошлым летом. Он называется Nanopore и базируется на очень интересной технологии, когда ДНК секвенируется путем пропускания через нанопору. Самый дешевый вариант Nanopore позиционируется как одноразовый домашний секвенатор и стоит $1000.
Третий прибор – PGM, полупроводниковый секвенатор, который стоит $50 000 у себя на родине и около 10 млн рублей (с доставкой, растаможиванием и т. д.) в России. Процесс секвенирования на нем занимает порядка нескольких часов.
Что ж, десяти миллионов у меня не было, а PGM захотелось. Пришлось сделать самому. Сначала вкратце о том, как происходит полупроводниковое секвенирование. Вся цепочка ДНК делится на фрагменты длиной по 300-400 нуклеотидов, называемые ридами. Затем риды прикрепляются к маленьким сферам и многократно копируются – в итоге на каждой сфере «висит» целый пучок одинаковых фрагментов ДНК. Копирование нужно для усиления сигнала от каждого конкретного рида. Набор разных сфер называется библиотекой ДНК.
Сердцем PGM является одноразовый чип – матрица, похожая на матрицу в фотокамере, только вместо пикселей, реагирующих на свет, здесь pH-транзисторы, реагирующие на изменение кислотно-щелочного баланса. Полученная библиотека ДНК загружается на чип, содержащий 10 млн лунок, на дне каждой из них находится pH-транзистор. В лунку умещается только одна сфера и, следовательно, риды только одного типа (с одной определенной последовательностью нуклеотидов). Далее на чип подаются реагенты таким образом, чтобы ДНК начала себя копировать. А копируется она линейно, то есть нуклеотиды прикрепляются к вновь создаваемой цепочке в том порядке, в котором они стоят в материнской цепочке. Поэтому на чип подается один тип нуклеотидов – и тут же фиксируется изменение pH в некоторых лунках (это значит, что в них произошло присоединение данного нуклеотида). Далее подается другой тип нуклеотидов и фиксируется изменение pH в лунках и т. д. Таким образом, подавая на чип все 4 типа нуклеотидов много раз, мы можем получить информацию о последовательности нуклеотидов в каждом риде. Затем математическими способами прочитанные короткие отрезки собираются на компьютере в единую цепочку. Чтобы собрать ее более-менее уверенно, каждый рид нужно прочесть примерно по 100 раз.
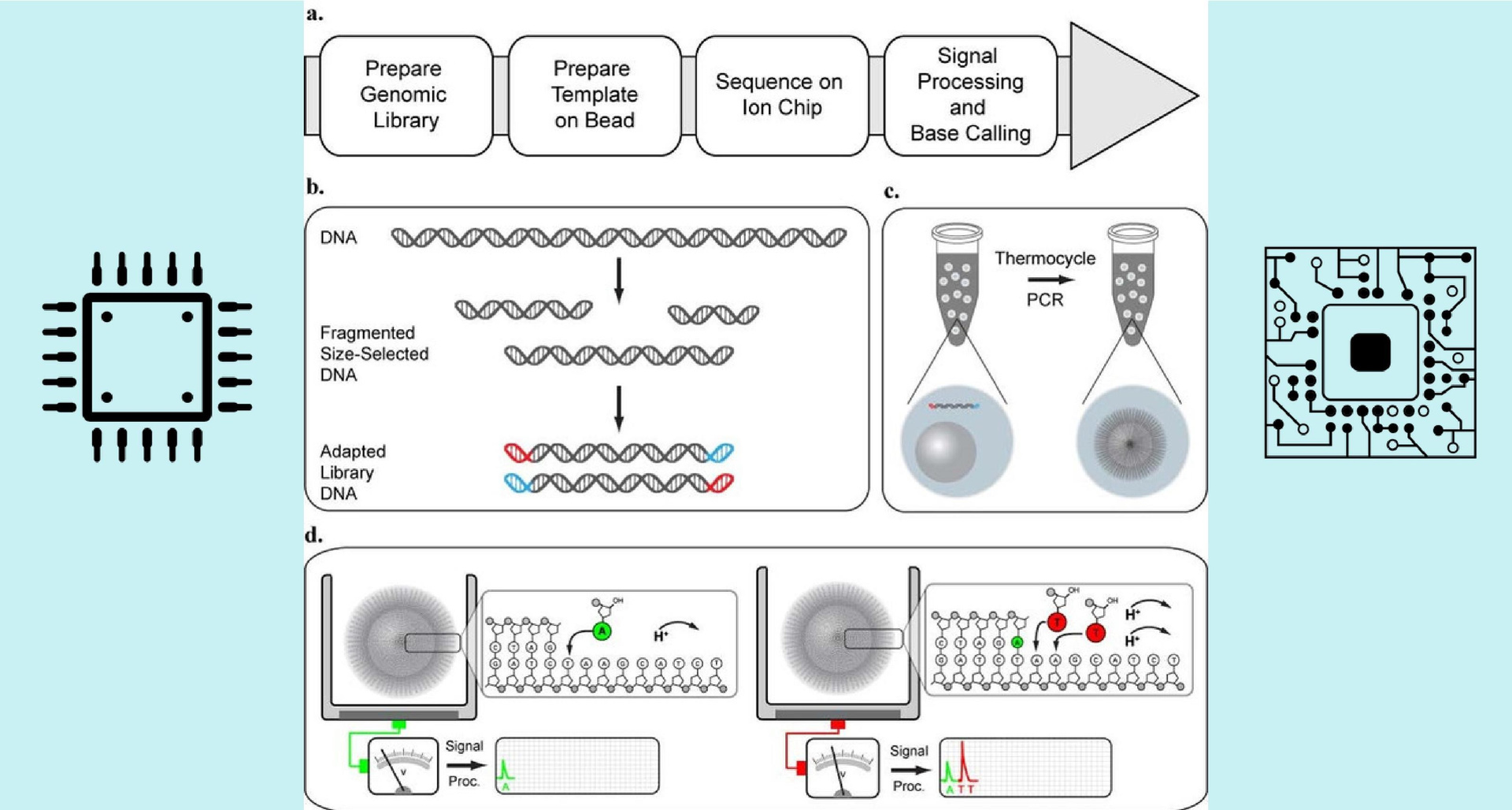
Рис.1. Полупроводниковое секвенирование
Теперь разберемся, из чего состоит сам прибор. Имеется, как мы уже знаем, чип, а также система подачи реагентов и материнская плата. Все секвенирование ведется именно на чипе – остальной аппарат только передает на него определенные сигналы, подает реагенты, считывает с него аналоговые сигналы, оцифровывает их и гонит полученный поток информации на компьютер, где данные накапливаются и обрабатываются.
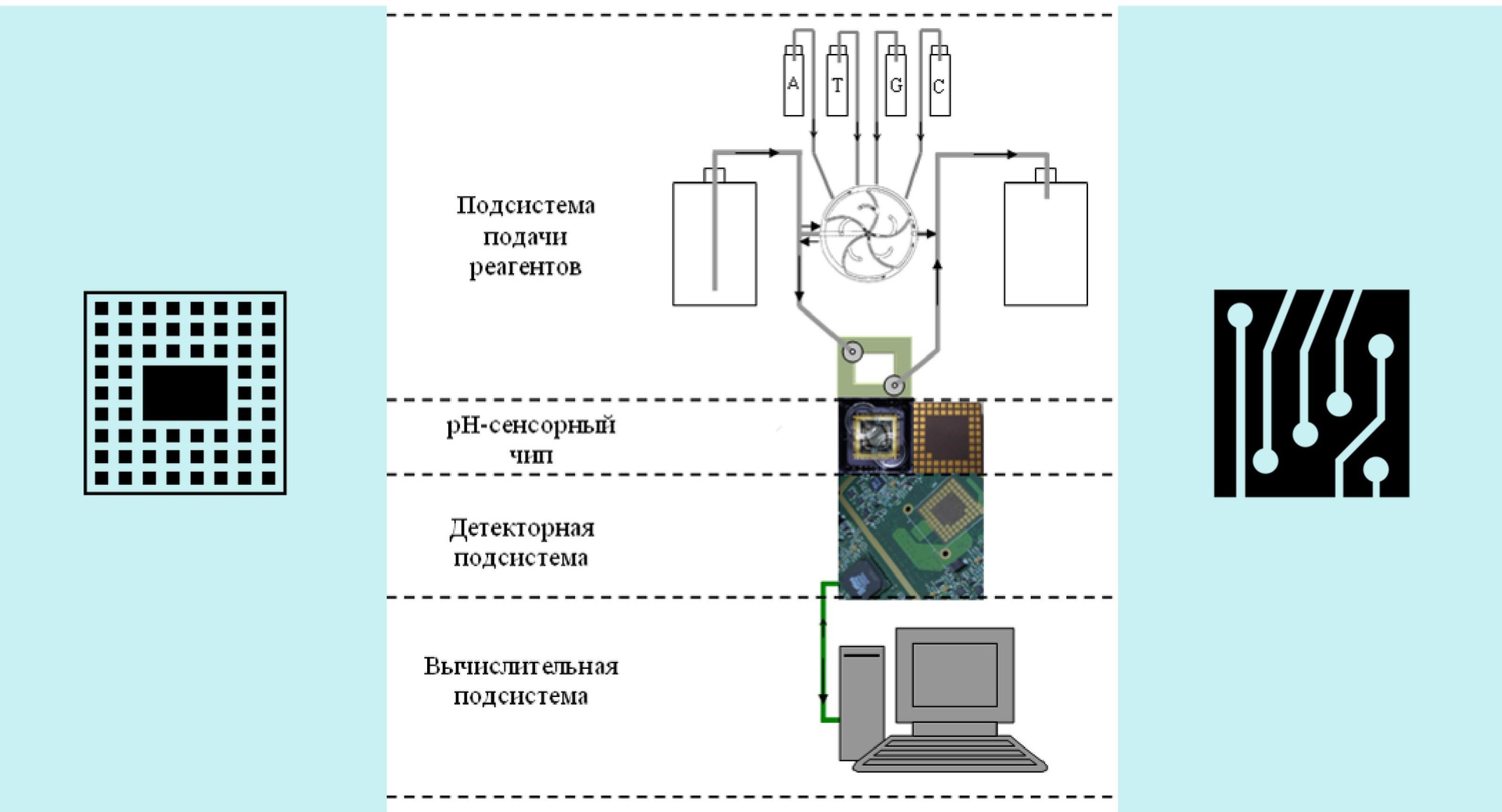
Рис. 2. Устройство секвенатора
Чип позиционируется как одноразовый и после использования выкидывается. Соответственно, там, где работает PGM, такие чипы можно достать бесплатно в любом количестве. Зачем их доставать, спросите вы? Дело в том, что чип мне уже удалось использовать многократно. По сути он вечен: достаточно хорошо промывать его – и можно применять вновь и вновь. По точности работы он ничем не будет отличаться от нового. Сама моя идея заключалась в том, чтобы сделать прибор под этот условно бесплатный чип.
Итак, передо мной встала задача реверс-инжиниринга чипа. Разумеется, никакой документации на заветную микросхему найти было нельзя – производитель не собирался делиться секретами производства, а хотел спокойно продавать свои приборы за $50 000. Для начала я сделал самое очевидное и простое: прозвонил контакты тестером. Стало ясно, где расположены цифровые и аналоговые входы-выходы, питание и прочее. Кое-какую информацию удалось почерпнуть из патентов на чип. Но всего этого, понятно, было недостаточно для создания полноценного продукта. Я еще повозился с чипом, проверял разные свои догадки, поэкспериментировал с подачей сигналов, но никуда принципиально не продвинулся. Пришлось поставить проект на паузу.

Рис. 3. Прозвонка чипа
А затем внезапно на Habrahabr мне попалась статья известного блоггера MonsterBars о том, как он делает реверс-инжиниринг чипов! Воодушевился, написал ему, написал другим энтузиастам, отправил запрос в Киев, где занимались фотографированием чипов. Из Киева ответили, что полировать по слоям они не умеют, могут только отснять верхний слой, а так как мой чип – многослойный, будет не понятно, куда идут дорожки от контактов. Потом познакомился с одним американцем, который тоже занимается реверс-инжинирингом чипов, послал ему свои микросхемы, но и тут дальше фотографирования верхнего слоя дело не пошло. Затем наткнулся в интернете на статью про тех, кто смог отреверсить чип Sony PlayStation и пр. («Слава героям!» и вот это все, если кто в курсе). Решил написать им с вопросами, нашел их ники – и тут же понял, что один из них мне знаком. Недавно товарищ свел меня со своим другом, который «тоже занимается генетикой на любительском уровне», мы пообщались с этим другом в Skype и на этом диалог закончили. И вот я понимаю, что мой новый приятель – мегакрутой мастер реверс-инжиниринга чипов. Тут же написал ему. Однако выяснилось, что, хоть помочь он и готов, у него нет микроскопа. Снова тупик.
А через несколько месяцев нужный микроскоп нашелся в соседней лаборатории! Правда, встроенная в него камера была ужасной, я фотографировал на мобильный телефон через окуляр и получал снимки вот такого качества:

Рис. 4. Чип под микроскопом
Затем на последний Новый год отличный микроскоп за 130 тыс. появился у меня на работе (я – специалист по квантовой криптографии). Мечты сбываются. Наконец, я смог нормально сфотографировать чип сверху.
Рис. 5. Мой рабочий микроскоп
А потом… Потом мне все-таки пришлось самому освоить технику его полировки. Трудность полировки заключается в том, чтобы снимать слои металла толщиной порядка 1 микрона – при этом ширина чипа составляет 1 сантиметр. Для сравнения скажу, что это примерно то же, что допустить на 1 км погрешность не более 10 см. Я очень старался. Результаты моих трудов представлены на следующем фото:

Рис. 6. Реверс-инжиниринг под оптическим микроскопом
Довольно хорошо видны нижний кремниевый слой, верхний слой с транзисторами, первый, второй, третий и четвертый слои металла.
Чип состоит из повторяющихся зон (типа сдвиговых регистров), и по таким картинкам было очень удобно его анализировать: сразу становилось ясно, что происходит на разных слоях. Я «отреверсил» самые «нафаршированные» участки с обилием логики, которые многократно повторялись. Но самым сложным оказалось отследить трассы, идущие по всему чипу, понять, какой внешний контакт к чему относится. С новогодних праздников до конца февраля, я, вооружившись новым прекрасным микроскопом, корпел над этой задачей – сидел на работе до десяти ночи, «реверсил», думал. И тут произошло новое чудо: товарищ смог организовать бесплатную фотосъемку чипа по слоям на электронном микроскопе в МИРЭА. «Фотосессия» крохи в 1 кв. см представляла собой 50 ГБ черно-белых фотографий.
Теперь все эти отдельные фотографии нужно было каким-то образом объединить в одну целую картинку. Чуть ли не в тот же день я написал на «питоне» программу, которая генерировала HTML-файл – при его открытии в браузере я получал требуемое. (Кстати, самая старая 10-я Opera справилась с этим лучше всего, рекомендую!) Затем на javascript написал еще одну программу, позволяющую сравнивать слои, плавно переходить между ними, выравнивать их, подбирать масштаб и т. д. Наконец, в моих руках были все инструменты для решения главных задач. Я отследил трассы, пронизывающие чип, и восстановил всю его структуру до последнего транзистора.
Еще одна фотография среза чипа, сделанная под рентгеном (в МИРЭА):
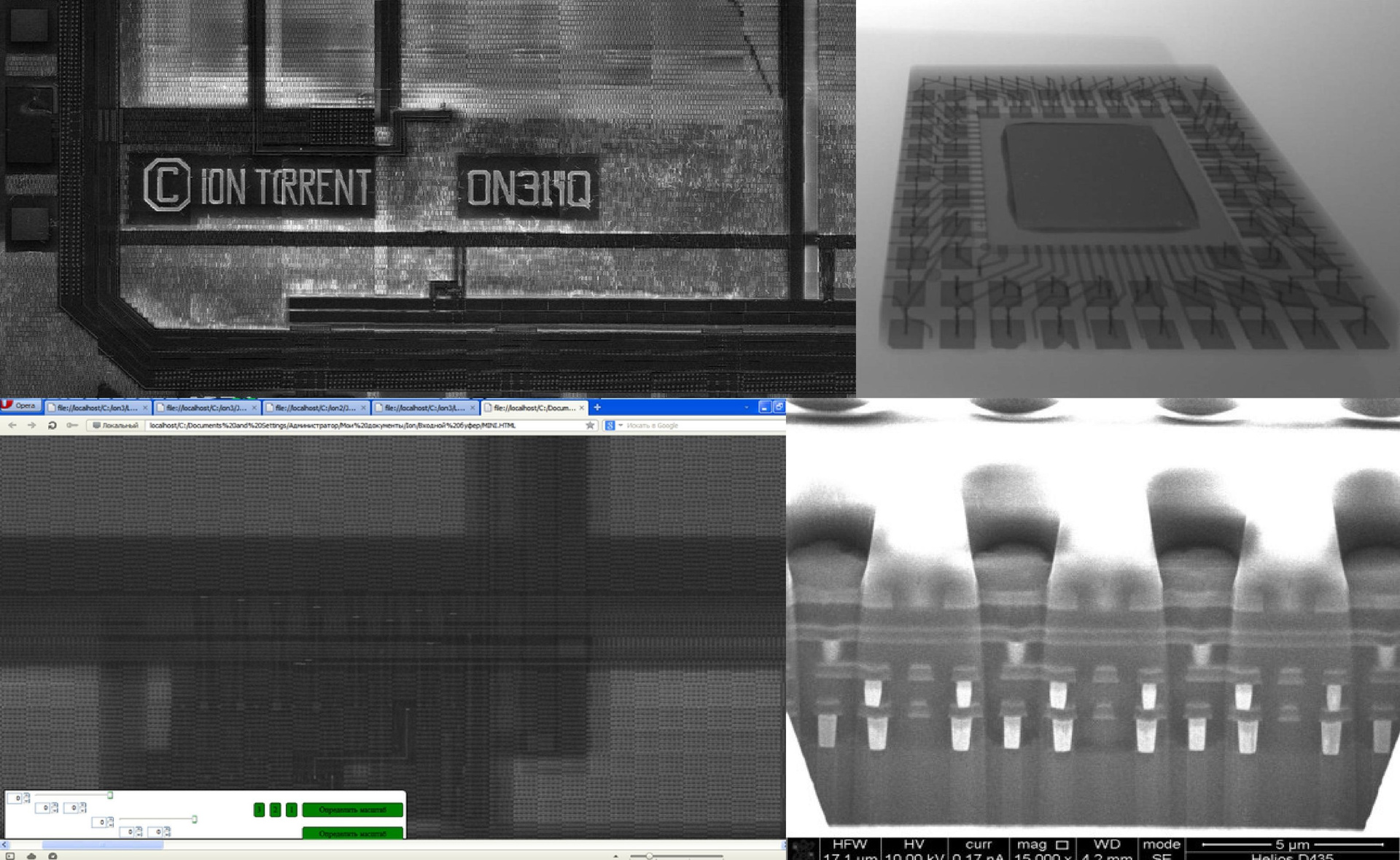
Рис. 7. Съемка под электронным микроскопом
Хорошо видны лунки, куда попадают сферы с ридами. Ниже располагаются три слоя металла, а еще ниже – слой с транзисторами.
Следующим этапом борьбы за светлое будущее стало создание под чип материнской платы. Спроектировал ее и отправил заказ на производство. А пока суд да дело использовал для работы с чипом плату «Марсоход-2» с FPGA. (FPGA – это, грубо говоря, массив из 10 000 универсальных логических элементов; программируя FPGA, мы можем получать любую логическую схему, легко обрабатывающую гигабитные потоки информации.) Прошивку для FPGA я написал сам, а кроме того, для динамического управления системой написал софт, который задает всю конфигурацию для FPGA. Потом вновь образовался полугодовой перерыв (я разводился, ездил в командировку на Байкал, готовил в лаборатории установку, которую демонстрировали Путину). Но в конце концов звезды сошлись: у меня появилось время, приехали готовые платы – и я собрал свою систему.
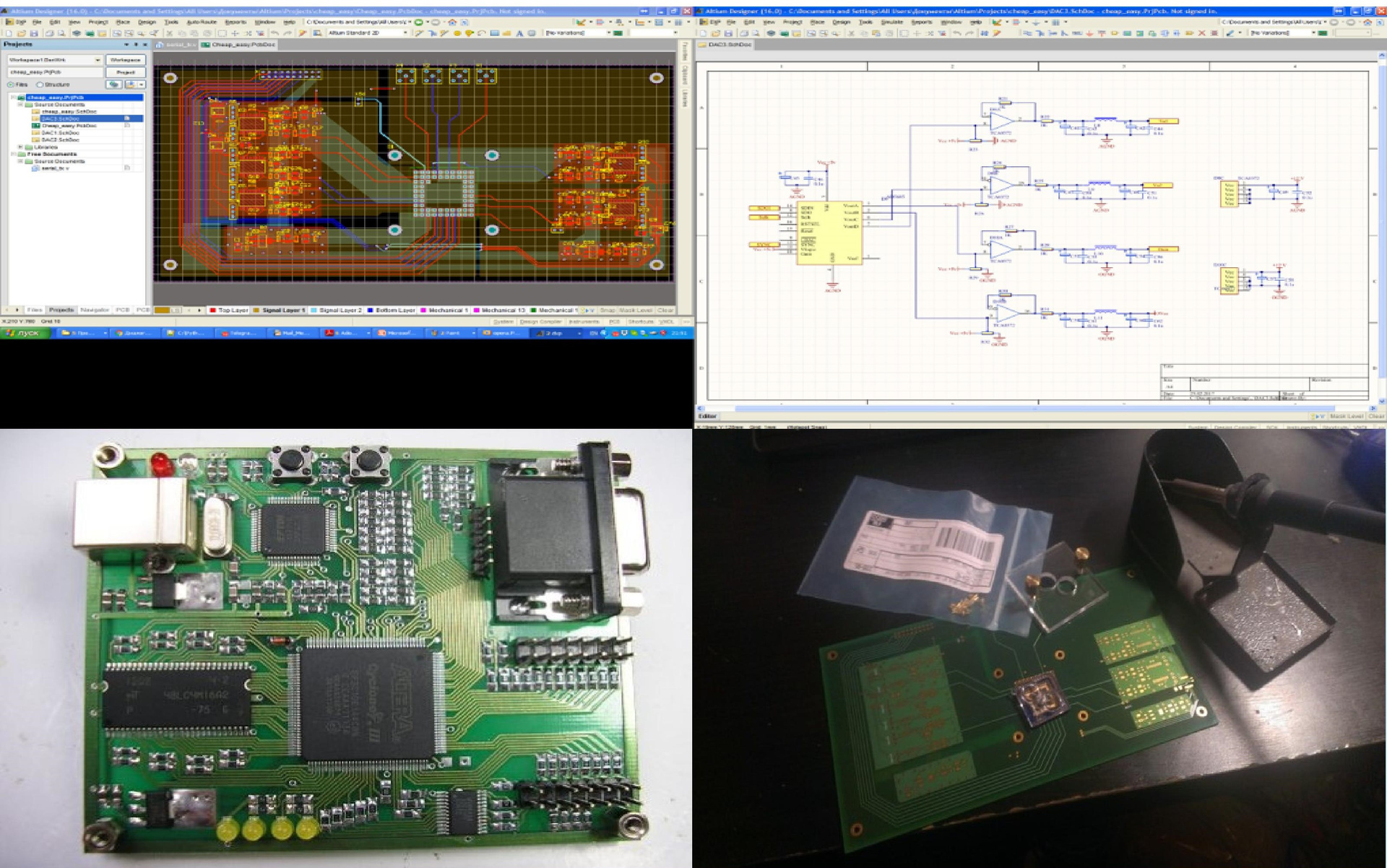
Рис. 8. Создание «железа»
Подал все необходимые сигналы и – о, чудо! – увидел на осциллографе сигнал с чипа. (Осциллограф я купил когда-то за 6 000 рублей на eBay, еще 1 000 стоила прошивка к нему.) На картинке хорошо видны пятна – капельки какого-то реагента.
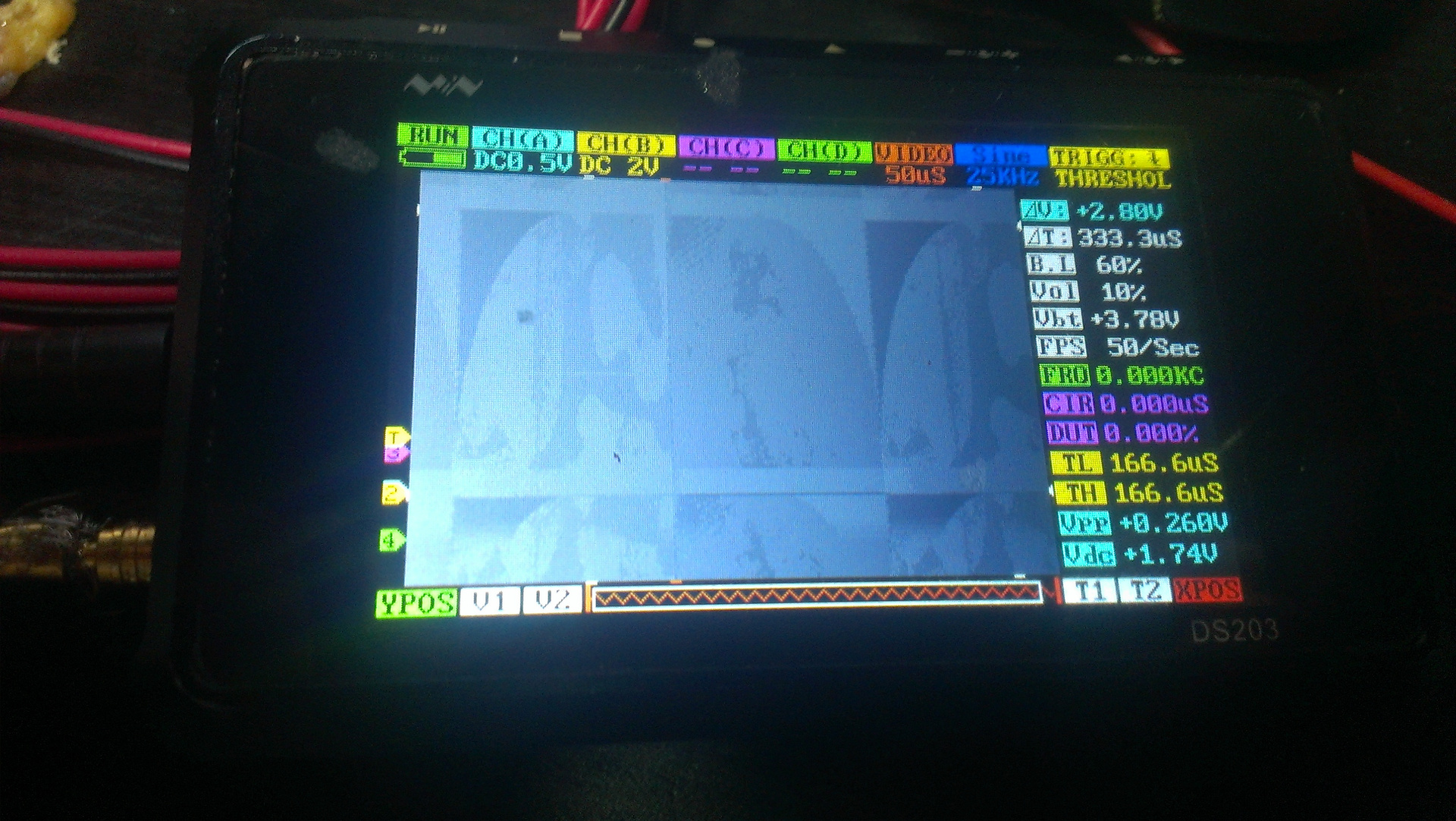
Рис. 9. Сигнал с чипа на осциллографе
Теперь мне нужно было придумать, как оцифровать эту картинку и передать ее на компьютер. Я собрал вот такую установку:
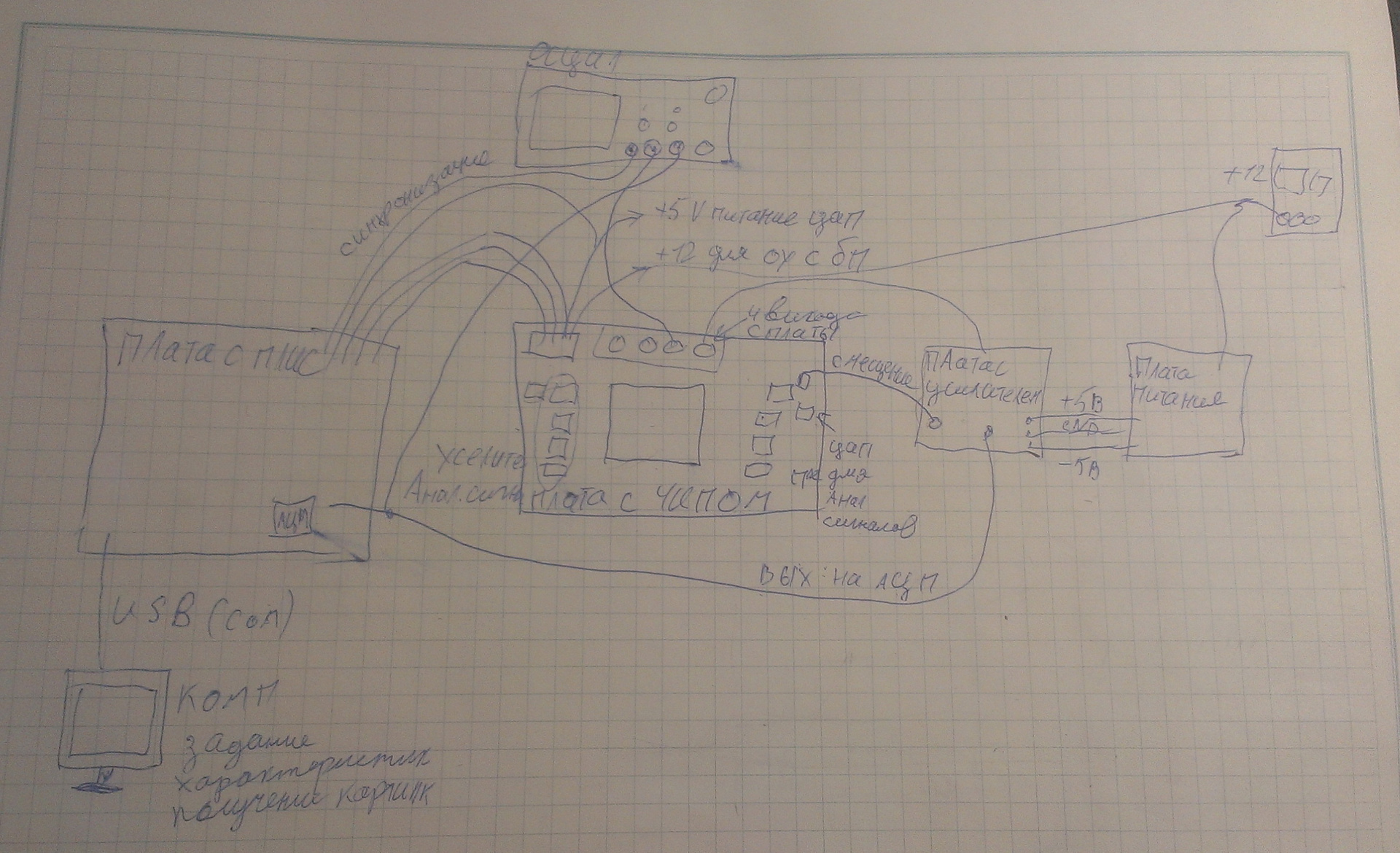
Рис.10. Схема прибора

Рис. 11. Готовая установка
Есть компьютер, который подает данные управления на плату с FPGA. Плата генерирует цифровые сигналы и отправляет их на чип. Сигнал с чипа идет на усилитель, далее – на АЦП на плате, оцифровывается и передается через COM-порт на компьютер. Вообще, пропускная способность COM-порта невелика: 15 килобит в секунду (т. к. в одном чипе находится от 1 млн до 10 млн «пикселей», а максимальная скорость передачи – 115200 бод). Тем не менее картинка на компьютер в итоге попадает.

Рис. 12. Обработанный сигнал на компьютере.
На фото выше видно, что, когда на использованный б/у-шный чип подается библиотека ДНК, чип заполняется неравномерно: по краям – в меньшей степени. Разные цвета обусловлены разным напряжением на pH-транзисторах. То есть мы можем ясно различить те лунки, куда попали сферы с ридами – впоследствии это поможет нам контролировать промывку чипа.
Соответственно, следующей задачей стала промывка чипа. Нужно было добиться, чтобы он стал, как новый. К счастью, у меня имелся совершенно новый чип в качестве референсного образца. На илл. А видно, что в активной области такой чип практически одного цвета (вертикальные повторяющиеся полосы – это просто шумы, наводки).

Рис. 13. Промывка чипа
На рис. 13 B неудачно промытый чип – он разноцветный. На рис.13 D – использованный, но хорошо промытый чип. Видно, что градиент по краям исчез. Тем не менее стоило бы еще доказать, что он действительно чистый и может использоваться повторно.
Поскольку библиотеки ДНК прикрепляются к танталовому покрытию чипа в кислой среде и открепляются – в щелочной (то есть при высоком pH), то чип промывается с помощью специальных полуавтоматических пипеток растворами с разными pH. На сегодняшний день мне удалось добиться практически полной очистки чипа.
У меня интересовались, почему, когда я полностью разобрался в структуре чипа, я не стал заказывать его изготовление, а предпочел по-прежнему искать и доставать б/у-шные, возиться с их промывкой и т. п. Да потому, что разработка микросхемы стоит огромных денег, миллионы долларов, и солидная часть этой суммы уходит на физическую отладку полученного продукта: подгонку, настройку всех параметров транзисторов и т. д. То есть просто скопировать логическую схему – недостаточно. Поэтому я беру условно бесплатную, уже готовую – спроектированную, изготовленную, отлаженную – микросхему и таким образом экономлю значительные средства, серьезно удешевляю проект.
Следующей моей задачей было собрать более продвинутый прибор, который позволял бы быстрее передавать информацию на компьютер и при этом не состоял бы из огромного количества отдельных плат.
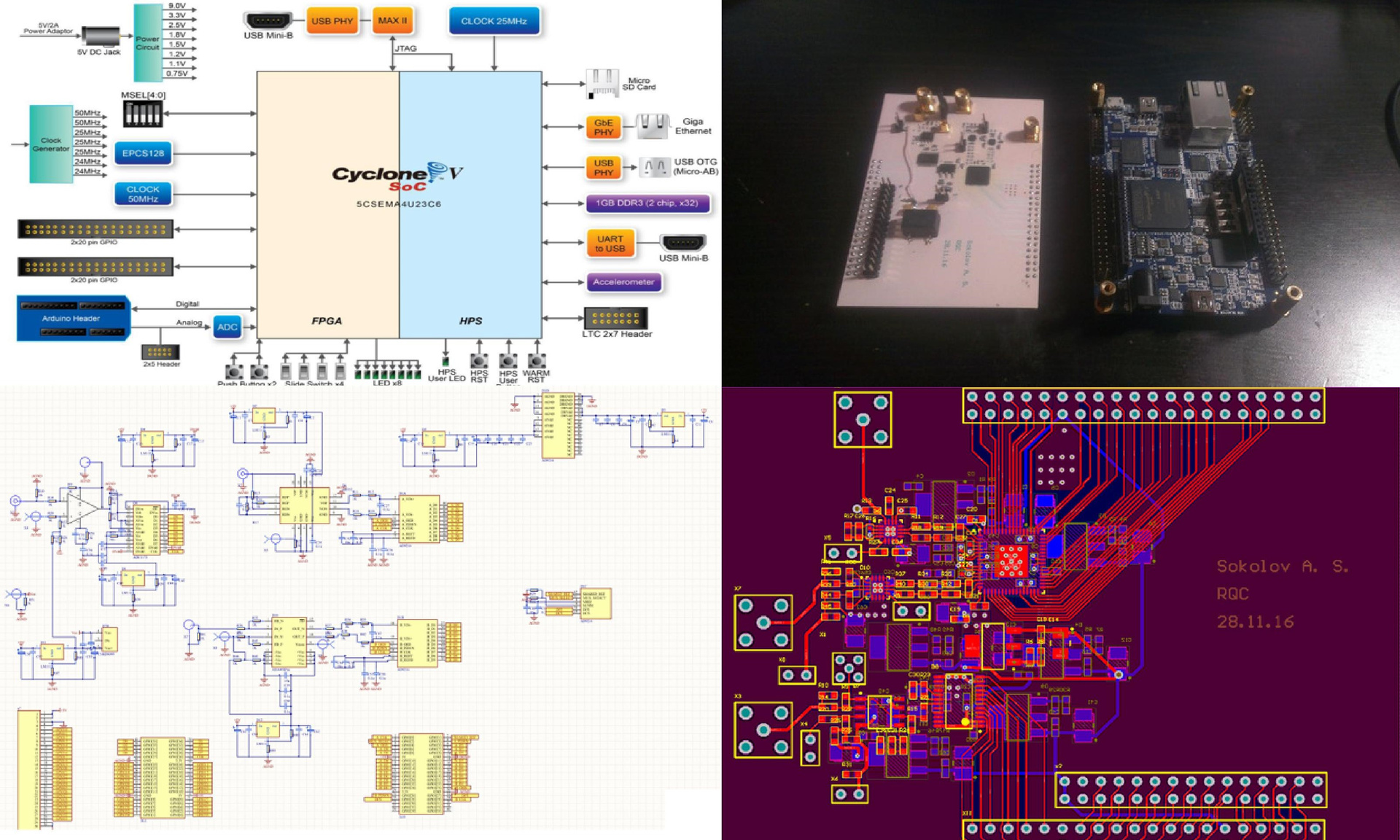
Рис.14 Разработка следующей версии прибора
Я взял новую плату с FPGA – на том же кристалле здесь было 2 ARM-ядра с Linux, имелся Gigabit Ethernet и прочие «плюшки», но зато, в отличие от предыдущего варианта, не было АЦП. Позже спроектировал еще одну плату, с высокоскоростными АЦП и всеми другими необходимыми элементами. Запустил – все заработало.
Что осталось сделать для появления финального прибора? Всего три вещи.
Первое. Нужен гигабитный интернет, быстрая передача данных на компьютер. Это я реализовал буквально вчера.
Второе. Система подачи реагентов. Проектирование специального клапана уже в процессе.
Третье. Софт для обработки информации с чипа. С ПО пока есть вопросы, поэтому приглашаю к сотрудничеству программистов.
Финальный прибор стоит 10 млн рублей. Себестоимость секвенирования составляет несколько тысяч долларов. Чипы обходятся от 100 до 1000 долларов – в зависимости от количества «пикселей» в них. (К слову, восстановление чипов само по себе может стать неплохим заработком, особенно учитывая, что для промывки нужно сделать лишь пару кликов.) Реагенты тоже покупаются, но в перспективе будут создаваться и они.
В общем все это очень интересно, но главное – за этим будущее. Сегодня биотехнологии занимают в мировом научно-техническом прогрессе то же место, что компьютерные технологии в 80-х гг. прошлого века. При этом секвенирование – одно из ключевых направлений для современной биологии и медицины. Ну и, конечно, биотехнологии – это очень прибыльно.
В последнее время на рынке появился полупроводниковый секвенатор S5, и в ближайшее будущие я планирую переключиться на него.
Буду рад пообщаться со всеми, кто захочет тем или иным образом поучаствовать в развитии этого проекта!
Спасибо за внимание!
Источник


